Les coronavirus (CoV) sont retrouvés dans de nombreuses espèces animales, en particulier les chauves-souris et les oiseaux, chez qui ils peuvent être responsables d’infections respiratoires, digestives et de troubles neurologiques. Jusqu’au début des années 2000, les coronavirus humains (HCoV) dits « classiques » (HCoV-229E, HCoV-HKU1, HCoV-NL63 et HCoV-OC43) n’étaient responsables que d’infections respiratoires saisonnières hautes et basses peu sévères chez l’immunocompétent. Récemment, des HCoV émergents, impliqués dans des infections respiratoires sévères et des troubles multi-organes, ont été identifiés chez l’homme : il s’agit des SARS-CoV-1 et -2 (2002 et 2019) [severe acute respiratory syndrome coronavirus] et du MERS-CoV (2012) [Middle East respiratory syndrome coronavirus].1
Généralités sur le SARS-CoV-2
Les CoV appartiennent à l’ordre des Nidovirales, à la famille des Coronaviridae et à la sous-famille des Orthocoronavirinae. Cette dernière est subdivisée en quatre genres (Alpha à Deltacoronavirus), auxquels appartiennent des CoV humains et animaux. Les HCoV « classiques » font partie des genres Betacoronavirus (HCoV-HKU1 et HCoV-OC43) et Alphacoronavirus (HCoV-229E et HCoV-NL63) ; les HCoV « émergents » appartiennent au genre Betacoronavirus (SARS-CoV-1, SARS-CoV-2 et MERS-CoV [tableau 1 ]).1, 2
Structure du virus et organisation génétique
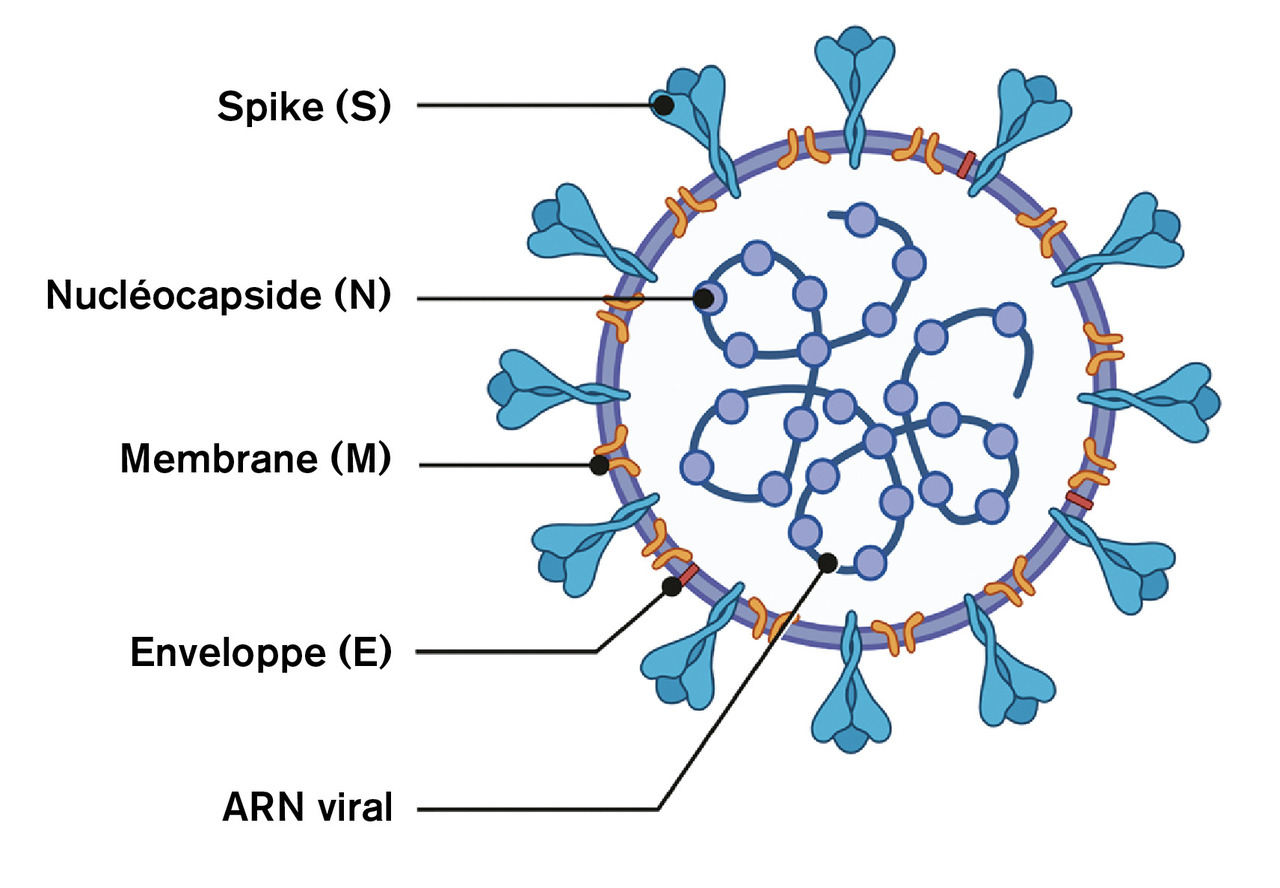
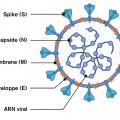
Les CoV sont des virus enveloppés à ARN monocaténaire linéaire non segmenté de polarité positive. La taille de la molécule d’ARN avoisine les 30 kilobases (kb) ; il s’agit du génome à ARN le plus grand qui ait été décrit chez un virus pathogène pour l’homme. L’organisation du génome du SARS-CoV-2 comprend deux régions non codantes (untranslated regions, ou UTR) en 5’ et 3’ et une partie, constituée de 10 à 12 ORF (open reading frame), codant pour des protéines structurales (dont la nucléoprotéine, la protéine Spike, les protéines de membrane et d’enveloppe) et pour des protéines non structurales impliquées notamment dans la réplication virale.1 L’ARN monocaténaire est encapsidé par la nucléoprotéine (N) formant la nucléocapside, l’ensemble étant protégé par l’enveloppe virale. L’enveloppe est composée par la protéine de membrane (M), la protéine d’enveloppe (E) et la protéine Spike (S) organisée en couronne à la surface de cette dernière (fig. 1 ).1
La protéine S est une protéine trimérique de fusion responsable de l’entrée du virus et portant des épitopes neutralisants.3 Elle possède un site de liaison spécifique appelé RBD (receptor binding domain) qui permet la reconnaissance de l’ACE2 (angiotensin-converting enzyme 2).3, 4 Les nombreuses mutations sur la protéine S sont à l’origine d’une grande diversité génétique, impliquées dans le franchissement de la barrière d’espèce et l’échappement aux anticorps neutralisants (monoclonaux thérapeutiques et polyclonaux vaccin-induits).5
La protéine S est une protéine trimérique de fusion responsable de l’entrée du virus et portant des épitopes neutralisants.3 Elle possède un site de liaison spécifique appelé RBD (receptor binding domain) qui permet la reconnaissance de l’ACE2 (angiotensin-converting enzyme 2).3, 4 Les nombreuses mutations sur la protéine S sont à l’origine d’une grande diversité génétique, impliquées dans le franchissement de la barrière d’espèce et l’échappement aux anticorps neutralisants (monoclonaux thérapeutiques et polyclonaux vaccin-induits).5
Tropisme cellulaire et cycle viral
Plusieurs récepteurs situés à la surface de la cellule épithéliale cible sont impliqués dans l’entrée du virus, dont le récepteur majeur ACE2 et la protéase TMPRSS2 (transmembrane protease serine 2) ;4 la distribution des récepteurs d’entrée sur la cellule cible détermine le tropisme viral. Le virus est alors endocyté, et les membranes virales et cellulaires fusionnent, permettant l’entrée du virus. L’ARN génomique est décapsidé et libéré dans le cytoplasme de la cellule infectée ; il est intégré dans un complexe de réplication et de transcription et transcrit en discontinu en ARN messagers (ARNm) subgénomiques grâce à l’ARN polymérase ARN-dépendante virale. Les ARNm sont ensuite traduits en protéines structurales et non structurales par les ribosomes cellulaires. Le virus est assemblé puis maturé successivement dans le réticulum endoplasmique puis l’appareil de Golgi. Enfin, les particules virales néoformées sont libérées de la cellule infectée par exocytose, en conservant l’intégrité cellulaire.
Modes de transmission
Le mode de transmission principal du SARS-CoV-2 est respiratoire, en majorité via les gouttelettes mais aussi par des particules mises en suspension dans l’air : virus résistant, il persiste dans les aérosols et peut ainsi se déposer sur certains objets (fomites) et persister sur certaines surfaces (jusqu’à plusieurs jours) ; ainsi la transmission indirecte liée aux fomites est-elle suspectée. D’autres modes de contamination accessoires sont évoqués (direct, vertical, fécal-oral, lié aux animaux domestiques...) ; la transmission respiratoire interhumaine reste néanmoins majoritaire.6
Contagiosité et dynamique de transmission
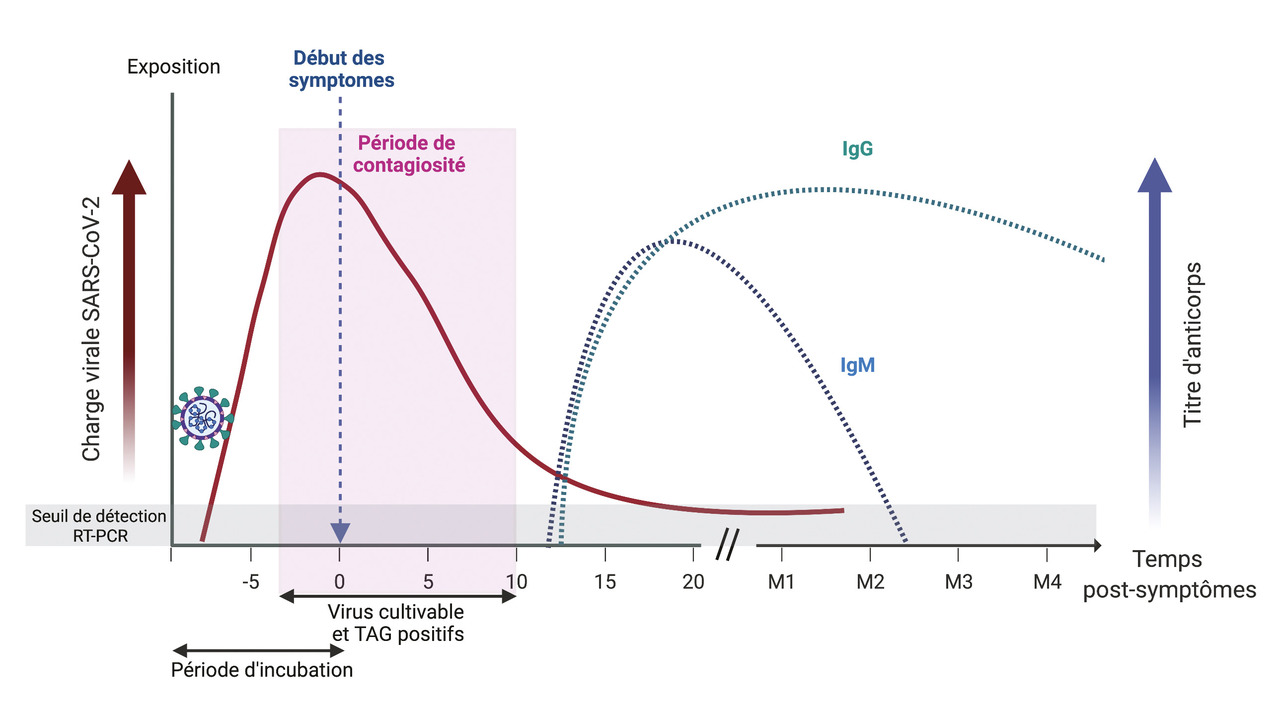
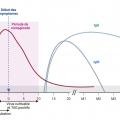
La contagiosité de l’individu infecté est corrélée à la charge virale. Pendant la période d’incubation estimée de cinq jours, l’individu est contagieux environ deux jours avant l’apparition des premiers symptômes, avec un pic d’infectiosité la veille des signes cliniques. En parallèle, le virus commence à répliquer dès vingt-quatre heures post-infection, et l’ARN viral est excrété dans les voies respiratoires supérieures, avec un pic de charge virale peu avant l’apparition des premiers symptômes (fig. 2 ).6 L’ARN viral peut persister plusieurs semaines, voire plusieurs mois chez des patients fortement immunodéprimés.
Certains individus sont asymptomatiques (environ 40 à 50 %) et constituent une source de la transmission.
Le taux de reproduction (R0), défini comme le nombre de cas secondaires générés par un cas index dans une population ni vaccinée ni immunisée, dépend du « fitness » du virus ; en effet, celui de la souche Wuhan (2019) était estimé à 2,5, alors que celui du variant Omicron atteindrait 10.7 Le R0 dépend également de l’état de contagiosité du cas index : certains ne transmettent pas le virus alors que d’autres, appelés « superspreaders », sont responsables de « clusters » de transmission, en particulier dans des lieux peu aérés.6
Certains individus sont asymptomatiques (environ 40 à 50 %) et constituent une source de la transmission.
Le taux de reproduction (R0), défini comme le nombre de cas secondaires générés par un cas index dans une population ni vaccinée ni immunisée, dépend du « fitness » du virus ; en effet, celui de la souche Wuhan (2019) était estimé à 2,5, alors que celui du variant Omicron atteindrait 10.7 Le R0 dépend également de l’état de contagiosité du cas index : certains ne transmettent pas le virus alors que d’autres, appelés « superspreaders », sont responsables de « clusters » de transmission, en particulier dans des lieux peu aérés.6
Diagnostic de l’infection
Pour affirmer le diagnostic, deux types de techniques peuvent être utilisés : recherche directe sur écouvillons naso- ou oropharyngés et recherche indirecte grâce aux sérologies.
Quelles techniques pour le diagnostic viral direct ?
Le virus est préférentiellement localisé dans les voies respiratoires supérieures, ce qui fait des écouvillons nasopharyngés (NP) ou oropharyngés des prélèvements non invasifs de choix. Le virus peut également être détecté dans des prélèvements respiratoires invasifs (liquide bronchoalvéolaire, liquide pleural), dans le sang et d’autres matrices en fonction des atteintes cliniques lors de l’infection (écouvillon rectal, liquide céphalorachidien…).8
Les tests diagnostiques doivent avoir une sensibilité élevée et une spécificité proche de 100 % ; leur performance dépend notamment du site de prélèvement, de sa qualité, de la symptomatologie du patient et du kit commercial utilisé.
L’Organisation mondiale de la santé (OMS) et la Haute Autorité de santé (HAS) recommandent que tout test diagnostiquant le SARS-CoV-2 ait des valeurs seuils minimales de sensibilité (supérieure ou égale à 80 %) et de spécificité (supérieure ou égale à 97 %) en comparaison au test de référence (RT-PCR NP [reverse transcription polymerase chain reaction], réaction de polymérisation en chaîne par transcription inverse, sur écouvillon nasopharyngé).9
Le diagnostic par RT-PCR sur prélèvement salivaire a été évalué, et plusieurs méta-analyses ont montré des performances comparables à celles du prélèvement nasopharyngé, avec une sensibilité estimée entre 83 et 95 %.13, 14 Les tests RT-PCR salivaires sont sensibles, simples, indolores et permettent l’autoprélèvement. Il s’agit d’une alternative de choix pour les patients « difficiles » à prélever (tels que les enfants) et pour les dépistages itératifs ; ils ont ainsi pu être déployés en 2021 dans les établissements scolaires et de santé sur recommandation de la HAS.15 Cependant, cette dernière a rappelé que si les tests RT-PCR salivaires venaient à se substituer aux tests nasopharyngés, ils pourraient omettre 10 à 20 % des sujets infectés par le SARS-CoV-2.9
Des tests antigéniques (TAG) ont été développés afin de raccourcir le délai de rendu : ils fournissent un résultat qualitatif (positif ou négatif) et rapide (en 15 à 30 minutes). Les TAG sur prélèvement nasopharyngé ont une sensibilité estimée entre 50 et 90 % et une spécificité de 90 à 100 %.13 Leur sensibilité est meilleure chez les patients symptomatiques et lorsque la charge virale est élevée (Ct inférieure ou égale à 25). La sensibilité des TAG sur autoprélèvement nasal (« autotests ») est médiocre chez la personne asymptomatique. La positivité des TAG se superpose avec la période de forte contagiosité ; le TAG peut être considéré comme un « test d’infectiosité » (fig. 2 ).
D’après l’OMS, les TAG nasopharyngés doivent être utilisés en priorité lorsque l’accès aux tests par RT-PCR est limité, chez les individus symptomatiques et chez certains individus asymptomatiques tels que les contacts de cas avérés et les agents de santé.10
Les tests diagnostiques doivent avoir une sensibilité élevée et une spécificité proche de 100 % ; leur performance dépend notamment du site de prélèvement, de sa qualité, de la symptomatologie du patient et du kit commercial utilisé.
L’Organisation mondiale de la santé (OMS) et la Haute Autorité de santé (HAS) recommandent que tout test diagnostiquant le SARS-CoV-2 ait des valeurs seuils minimales de sensibilité (supérieure ou égale à 80 %) et de spécificité (supérieure ou égale à 97 %) en comparaison au test de référence (RT-PCR NP [reverse transcription polymerase chain reaction], réaction de polymérisation en chaîne par transcription inverse, sur écouvillon nasopharyngé).9
Amplification des acides nucléiques : la référence
La technique de référence repose sur la détection de l’ARN viral du SARS-CoV-2 par techniques d’amplification des acides nucléiques (TAAN) sur écouvillon nasopharyngé. Les TAAN validées en France sont la RT-PCR, la RT-LAMP (amplification isothermique sans extraction d’ARN) et la TMA (transcription médiée par l’amplification). À l’exception des appareils délocalisés déployés dans certains services hospitaliers d’urgence (technique RT-LAMP), le diagnostic par TAAN s’effectue dans un laboratoire de biologie médicale. La technique est automatisée ou semi-automatisée, et le délai de rendu des résultats par le biologiste peut être de 45 minutes à plusieurs heures, selon la technologie utilisée. Les kits de détection de l’ARN viral par RT-PCR doivent disposer d’un marquage « CE » IVD (in vitro diagnostics) et cibler au moins deux gènes viraux.11 Le résultat est qualitatif (positif/négatif) ,et certains auto–mates de RT-PCR en temps réel fournissent une estimation semi-quantitative de l’ARN viral grâce aux valeurs de Ct (cycle threshold). D’après les recommandations de la Société française de microbiologie, si la valeur de Ct est inférieure ou égale à 33, la présence d’ARN détecté est compatible avec une excrétion virale significative, valeur à adapter à la technique utilisée.12Prélèvements salivaires et tests antigéniques, des alternatives ?
Le prélèvement nasopharyngé, à risque importantd’aérosolisation, requiert un personnel qualifié et l’utilisation d’un équipement adapté. Enfin, il peut s’avérer douloureux et difficilement réalisable chez certains patients (enfants, patients non coopératifs) et inconfortable lors des dépistages itératifs (patients hospitalisés, personnel soignant, collectivités). Pour pallier ces inconvénients et les délais de rendu parfois longs de la RT-PCR, des alternatives ont été étudiées.Le diagnostic par RT-PCR sur prélèvement salivaire a été évalué, et plusieurs méta-analyses ont montré des performances comparables à celles du prélèvement nasopharyngé, avec une sensibilité estimée entre 83 et 95 %.13, 14 Les tests RT-PCR salivaires sont sensibles, simples, indolores et permettent l’autoprélèvement. Il s’agit d’une alternative de choix pour les patients « difficiles » à prélever (tels que les enfants) et pour les dépistages itératifs ; ils ont ainsi pu être déployés en 2021 dans les établissements scolaires et de santé sur recommandation de la HAS.15 Cependant, cette dernière a rappelé que si les tests RT-PCR salivaires venaient à se substituer aux tests nasopharyngés, ils pourraient omettre 10 à 20 % des sujets infectés par le SARS-CoV-2.9
Des tests antigéniques (TAG) ont été développés afin de raccourcir le délai de rendu : ils fournissent un résultat qualitatif (positif ou négatif) et rapide (en 15 à 30 minutes). Les TAG sur prélèvement nasopharyngé ont une sensibilité estimée entre 50 et 90 % et une spécificité de 90 à 100 %.13 Leur sensibilité est meilleure chez les patients symptomatiques et lorsque la charge virale est élevée (Ct inférieure ou égale à 25). La sensibilité des TAG sur autoprélèvement nasal (« autotests ») est médiocre chez la personne asymptomatique. La positivité des TAG se superpose avec la période de forte contagiosité ; le TAG peut être considéré comme un « test d’infectiosité » (
D’après l’OMS, les TAG nasopharyngés doivent être utilisés en priorité lorsque l’accès aux tests par RT-PCR est limité, chez les individus symptomatiques et chez certains individus asymptomatiques tels que les contacts de cas avérés et les agents de santé.10
Diagnostic indirect : la sérologie
La production d’anticorps spécifiques dirigés contre le SARS-CoV-2 commence, en général, après la première semaine d’infection ; la majorité des patients développent des immunoglobulines spécifiques (IgG et IgM) en dix à quinze jours, qui sont détectables par les méthodes sérologiques. Les IgG persistent alors que le taux d’IgM diminue dès le premier mois (fig. 2 ).16, 17
Deux techniques sérologiques sont utilisées : les tests automatisés immuno-enzymatiques de type ELISA (enzyme-linked immunosorbent assay) ou CLIA (chemiluminescence immunoassay) et les tests immuno-chromatographiques unitaires. Il existe de multiples trousses commerciales utilisant plusieurs types d’antigènes ; cela pose un problème d’harmonisation.17 Les antigènes utilisés sont la protéine S et/ou la protéine N. La présence d’IgG anti-N témoigne d’une infection naturelle, mais leur taux sérique décroît en quelques mois ; la présence d’IgG anti-S témoigne d’une infection naturelle et/ou d’une vaccination et elle persiste plus longtemps. Le résultat d’IgG anti-S est standardisé et rendu en BAU/mL (binding antibody units par millilitre [unités OMS]) à l’aide d’un facteur de conversion propre à chaque fournisseur.
Pour augmenter la performance des techniques, il est recommandé de combiner la recherche des anticorps anti-N et anti-S.16
Les tests sérologiques sont indiqués notamment en diagnostic initial et de rattrapage chez les patients symptomatiques graves hospitalisés ayant des signes cliniques évocateurs et une RT-PCR négative. Chez les individus immunodéprimés n’ayant pas répondu à la vaccination, le seuil de 264 BAU/mL est admis par la Direction générale de la santé comme taux en dessous duquel il faut proposer un traitement par anticorps monoclonaux en prophylaxie primaire ou post-exposition.18 En revanche, du fait de l’évolution de l’épidémie, des différentes techniques utilisées et des seuils variés d’interprétation de la séropositivité, il n’est pas possible à ce jour de définir une valeur-seuil de taux d’anticorps protecteurs. Pour évaluer la protection, il faudrait mesurer le taux d’anticorps neutralisants par des tests de neutralisation utilisés en recherche et étudier l’éventuelle corrélation entre la sévérité de la maladie et un seuil d’immunoneutralisation ; plusieurs tests de neutralisation, plus accessibles pour un potentiel usage en routine, sont en cours de développement.[17]
Deux techniques sérologiques sont utilisées : les tests automatisés immuno-enzymatiques de type ELISA (enzyme-linked immunosorbent assay) ou CLIA (chemiluminescence immunoassay) et les tests immuno-chromatographiques unitaires. Il existe de multiples trousses commerciales utilisant plusieurs types d’antigènes ; cela pose un problème d’harmonisation.17 Les antigènes utilisés sont la protéine S et/ou la protéine N. La présence d’IgG anti-N témoigne d’une infection naturelle, mais leur taux sérique décroît en quelques mois ; la présence d’IgG anti-S témoigne d’une infection naturelle et/ou d’une vaccination et elle persiste plus longtemps. Le résultat d’IgG anti-S est standardisé et rendu en BAU/mL (binding antibody units par millilitre [unités OMS]) à l’aide d’un facteur de conversion propre à chaque fournisseur.
Pour augmenter la performance des techniques, il est recommandé de combiner la recherche des anticorps anti-N et anti-S.16
Les tests sérologiques sont indiqués notamment en diagnostic initial et de rattrapage chez les patients symptomatiques graves hospitalisés ayant des signes cliniques évocateurs et une RT-PCR négative. Chez les individus immunodéprimés n’ayant pas répondu à la vaccination, le seuil de 264 BAU/mL est admis par la Direction générale de la santé comme taux en dessous duquel il faut proposer un traitement par anticorps monoclonaux en prophylaxie primaire ou post-exposition.18 En revanche, du fait de l’évolution de l’épidémie, des différentes techniques utilisées et des seuils variés d’interprétation de la séropositivité, il n’est pas possible à ce jour de définir une valeur-seuil de taux d’anticorps protecteurs. Pour évaluer la protection, il faudrait mesurer le taux d’anticorps neutralisants par des tests de neutralisation utilisés en recherche et étudier l’éventuelle corrélation entre la sévérité de la maladie et un seuil d’immunoneutralisation ; plusieurs tests de neutralisation, plus accessibles pour un potentiel usage en routine, sont en cours de développement.[17]
Variants du SARS-CoV-2
Des mutations sur le génome viral du SARS-CoV-2 ont été identifiées dès le début de la pandémie, et des variants ont émergé lors de l’été 2020.
Plasticité du génome
La majorité des mutations ont peu d’incidence sur les propriétés du virus. Mais certaines sont sélectionnées car elles sont associées à un meilleur « fitness » viral (augmentation de la réplication et de la transmissibilité) qui permet l’échappement à la réponse immunitaire adaptative ou modifie les interactions entre le virus et les protéines de la cellule hôte.
L’évolution du génome viral est préférentiellement liée à des mutations ponctuelles et à des phénomènes d’insertions/délétions.5 Les mutations ponctuelles sont rares (environ 10-6 par nucléotide et par cycle de réplication) car le virus possède une exonucléase qui corrige les erreurs d’incorporation de l’ARN polymérase virale ; elles ne permettent pas au virus d’évoluer rapidement. En revanche, dès lors que les mutations ou les événements d’insertions/délétions se produisent dans la région codant pour la protéine S, cela impacte fortement les propriétés du virus.19
Trois hypothèses sur l’origine d’apparition des variants sont avancées :
– l’évolution du génome viral serait due à des recombinaisons homologues entre le génome du SARS-CoV-2 et celui d’autres coronavirus ;5
– le « patient zéro » serait un hôte immunodéprimé chez lequel le virus aurait persisté pendant plusieurs mois ; des mutations se seraient ainsi accumulées et celles conférant un avantage sélectif auraient été sélectionnées ;20
– l’homme serait un réservoir infectant pour l’animal (« rétrozoonose »). Il s’agirait d’une infection « reverse » ; les mutations d’adaptation du virus à son nouvel hôte animal conduiraient à un variant significativement différent qui pourrait ensuite réinfecter l’homme.21, 22
L’évolution du génome viral est préférentiellement liée à des mutations ponctuelles et à des phénomènes d’insertions/délétions.5 Les mutations ponctuelles sont rares (environ 10-6 par nucléotide et par cycle de réplication) car le virus possède une exonucléase qui corrige les erreurs d’incorporation de l’ARN polymérase virale ; elles ne permettent pas au virus d’évoluer rapidement. En revanche, dès lors que les mutations ou les événements d’insertions/délétions se produisent dans la région codant pour la protéine S, cela impacte fortement les propriétés du virus.19
Trois hypothèses sur l’origine d’apparition des variants sont avancées :
– l’évolution du génome viral serait due à des recombinaisons homologues entre le génome du SARS-CoV-2 et celui d’autres coronavirus ;5
– le « patient zéro » serait un hôte immunodéprimé chez lequel le virus aurait persisté pendant plusieurs mois ; des mutations se seraient ainsi accumulées et celles conférant un avantage sélectif auraient été sélectionnées ;20
– l’homme serait un réservoir infectant pour l’animal (« rétrozoonose »). Il s’agirait d’une infection « reverse » ; les mutations d’adaptation du virus à son nouvel hôte animal conduiraient à un variant significativement différent qui pourrait ensuite réinfecter l’homme.21, 22
Classifications des génomes : l’alphabet grec pour l’OMS
Les classifications des génomes du SARS-CoV-2 sont fondées sur les différentes mutations qui confèrent un avantage sélectif aux variants. Actuellement, plusieurs systèmes de nomenclature permettent de suivre les lignées génétiques du SARS-CoV-2 : GISAID (Global initiative on sharing all influenza data), Nextstrain et Pango.5
L’OMS a établi une nomenclature plus accessible au grand public : à chaque nouveau variant est attribuée une lettre de l’alphabet grec. Les variants sont classés en différentes catégories en fonction de leurs caractères phénotypiques, et l’OMS a ainsi défini les variants à suivre (variant of interest, VoI) et les variants préoccupants (variant of concern, VoC) [tableau 2 ].23 Les VoC (Alpha, Bêta, Gamma, Delta et Omicron) peuvent avoir une transmissibilité et une pathogénicité augmentées, être moins efficacement neutralisés par les anticorps après infection et/ou vaccination et être moins bien détectés par les méthodes diagnostiques.23
L’OMS a établi une nomenclature plus accessible au grand public : à chaque nouveau variant est attribuée une lettre de l’alphabet grec. Les variants sont classés en différentes catégories en fonction de leurs caractères phénotypiques, et l’OMS a ainsi défini les variants à suivre (variant of interest, VoI) et les variants préoccupants (variant of concern, VoC) [
Le variant Omicron
Le VoC Omicron présente plus de 50 mutations par rapport à la souche Wuhan, dont plus de 30 sont situées sur la protéine S.24 Certaines mutations sont situées au sein du domaine RBD (receptor binding domain) de la protéine S, augmentant son affinité pour le récepteur ACE2. De plus, le virus échappe au système immunitaire car les anticorps neutralisants ciblent principalement le motif RBM (receptor binding motif) au sein du RBD ; cela impacte l’efficacité des anticorps polyclonaux post-vaccinaux et celle d’une majorité d’anticorps monoclonaux thérapeutiques ; seuls ceux ciblant un épitope extérieur au RBM restent actifs.
En revanche, il est décrit que l’affinité des anticorps dirigés contre Omicron est augmentée par des expositions répétées au virus (infections/boosts vaccinaux).24
En revanche, il est décrit que l’affinité des anticorps dirigés contre Omicron est augmentée par des expositions répétées au virus (infections/boosts vaccinaux).24
Surveillance des variants
Santé publique France a déployé un système de surveillance des variants du SARS-CoV-2 grâce à deux méthodes : le criblage et le séquençage.
Le criblage est une technique de RT-PCR permettant d’identifier des mutations ciblées faisant suspecter un variant. Les résultats sont transmis en temps réel à Santé publique France via le Système d’information pour la déclaration des essais de produits (SIDEP) pour permettre le suivi épidémiologique. La stratégie est remodelée régulièrement en fonction de l’évolution de l’épidémie.25 Parmi les mutations d’intérêt recherchées, la N501Y contribue à augmenter le « fitness » viral, et les mutations E484K et K417N permettent au virus d’échapper au système immunitaire.19
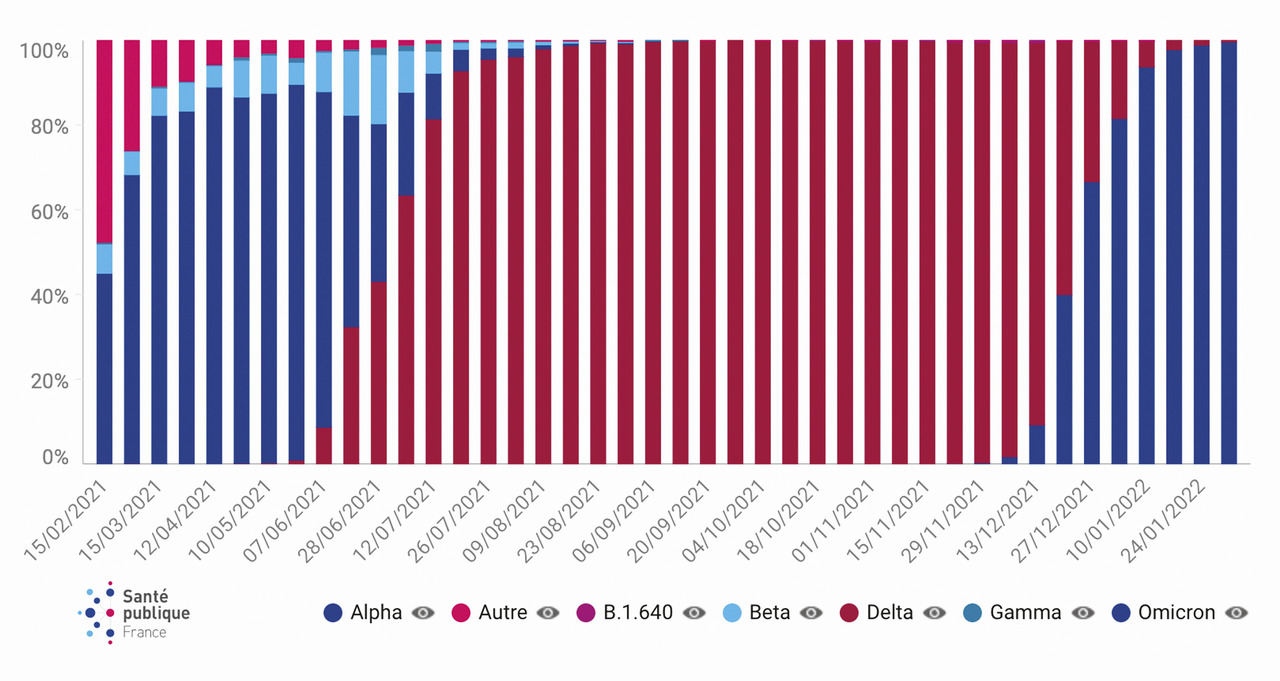
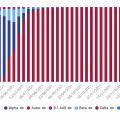
Enfin, le séquençage profond du génome complet viral est la méthode de référence permettant de mettre en évidence un nouveau variant. Ces séquences alimentent les « enquêtes Flash » hebdomadaires mises en place depuis janvier 2021 par Santé publique France, qui cartographient, à un instant donné, les variants circulant sur le territoire français. Ces enquêtes ont mis en évidence en août 2021 la disparition du variant Alpha au profit de Delta puis, en janvier 2022, l’émergence d’Omicron du fait de l’avantage sélectif conféré par ses nombreuses mutations (fig. 3 ).25 Avec l’arrivée d’Omicron, le criblage est devenu un examen indispensable en complément de la sérologie, avant l’instauration d’un traitement par anticorps monoclonaux afin d’adapter la stratégie thérapeutique au variant.
Le criblage est une technique de RT-PCR permettant d’identifier des mutations ciblées faisant suspecter un variant. Les résultats sont transmis en temps réel à Santé publique France via le Système d’information pour la déclaration des essais de produits (SIDEP) pour permettre le suivi épidémiologique. La stratégie est remodelée régulièrement en fonction de l’évolution de l’épidémie.25 Parmi les mutations d’intérêt recherchées, la N501Y contribue à augmenter le « fitness » viral, et les mutations E484K et K417N permettent au virus d’échapper au système immunitaire.19
Enfin, le séquençage profond du génome complet viral est la méthode de référence permettant de mettre en évidence un nouveau variant. Ces séquences alimentent les « enquêtes Flash » hebdomadaires mises en place depuis janvier 2021 par Santé publique France, qui cartographient, à un instant donné, les variants circulant sur le territoire français. Ces enquêtes ont mis en évidence en août 2021 la disparition du variant Alpha au profit de Delta puis, en janvier 2022, l’émergence d’Omicron du fait de l’avantage sélectif conféré par ses nombreuses mutations (
Des tests au service du contrôle de l’épidémie
Depuis l’émergence de l’épidémie de SARS-CoV-2 fin 2019, la recherche a permis d’acquérir de très nombreuses connaissances sur le virus. Le développement des tests a permis d’adapter nos stratégies diagnostiques et de dépistage à large échelle pour un meilleur contrôle de l’épidémie.
Références
1. Tang D, Comish P, Kang R. The hallmarks of COVID-19 disease. Hobman TC, éditeur. PLOS Pathog [Internet]. mai 2020 [consulté le 7 février 2022]; Disponible sur : https://doi.org/10.1371/journal.ppat.1008536
2. Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. The species severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol 2020;5(4):536‑44.
3. Walls AC, Park Y-J, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell 2020;180:281‑92.
4. Jackson CB, Farzan M, Chen B, Choe H. Mechanisms of SARS-CoV-2 entry into cells. Nat Rev Mol Cell Biol 2022;23(1):3‑20.
5. Amoutzias GD, Nikolaidis M, Tryfonopoulou E, Chlichlia K, Markoulatos P, Oliver SG. The remarkable evolutionary plasticity of coronaviruses by mutation and recombination: insights for the COVID-19 pandemic and the future evolutionary paths of SARS-CoV-2. Viruses [Internet]. Janvier 2022 [consulté le 1er février 2022]. Disponible sur : https://doi.org/10.3390/v14010078
6. Meyerowitz EA, Richterman A, Gandhi RT, Sax PE. Transmission of SARS-CoV-2: a review of viral, host, and environmental factors. Ann Intern Med 2021;174(1):69‑79.
7. Burki TK. Omicron variant and booster COVID-19 vaccines. Lancet Respir Med [Internet]. Février 2022 [consulté le 23 février 2022]; Disponible sur : https://doi.org/10.1016/S2213-2600(21)00559-2
8. Bwire GM, Majigo MV, Njiro BJ, Mawazo A. Detection profile of SARS-CoV-2 using RT-PCR in different types of clinical specimens: A systematic review and meta-analysis. J Med Virol 2021;93(2):719‑25.
9. Haute Autorité de santé. Méta-analyse de l’intérêt diagnostique des tests RT-PCR salivaires de détection du SARS- CoV-2 - 10 février 2021 [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2021-02/meta-analyse_rt-pcr_salive_vd.pdf
10. Organisation mondiale de la santé. Détection des antigènes pour le diagnostic de l’infection à SARS-CoV-2 - 6 octobre 2020 [Internet]. Disponible sur : https://apps.who.int/iris/bitstream/handle/10665/347019/WHO-2019-nCoV-Antigen-Detection-2021.1-fre.pdf
11. Haute Autorité de santé. Avis n° 2020.0020/AC/SEAP du 6 mars 2020 : détection du génome du coronavirus SARS-CoV-2 par technique de transcription inverse suivie d’une amplification [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2020-03/ac_rt-pcr_sars_cov2_cd_20200306_visasj_v2_post_cd.pdf
12. Société française de dermatologie. Avis du 25 septembre 2020 de la Société française de microbiologie relatif à l’interprétation de la valeur de Ct (estimation de la charge virale) obtenue en cas de RT-PCR SARS-CoV-2 positive sur les prélèvements cliniques réalisés à des fins diagnostiques ou de dépistage, version 4 [Internet]. Disponible sur: https://www.sfm-microbiologie.org/wp-content/uploads/2021/01/Avis-SFM-valeur-Ct-excrétion-virale-_-Version-def-14012021_V4.pdf
13. Kernéis S, Elie C, Fourgeaud J, Choupeaux L, Mercier Delarue S, Alby M-L, et al. Accuracy of saliva and nasopharyngeal sampling for detection of SARS-CoV-2 in community screening. Eur J Clin Microbiol 2021;40(11):2379‑88.
14. Butler-Laporte G, Lawandi A, Schiller I, Yao M, Dendukuri N, McDonald EG, et al. Comparison of saliva and nasopharyngeal swab nucleic acid amplification testing for detection of SARS-CoV-2: a systematic review and meta-analysis. JAMA Intern Med 2021;181(3):353‑60.
15. Haute Autorité de santé. Avis n° 2021.0007/AC/SEAP du 10 février 2021 : Détection du génome du virus SARS-CoV-2 par technique de transcription inverse suivie d’une amplification (RT-PCR) sur prélèvement salivaire [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2021-02/ac_2021_0007_rt-pcr_salivaire_covid-19.pdf
16. Caruana G, Croxatto A, Coste AT, Opota O, Lamoth F, Jaton K, et al. Diagnostic strategies for SARS-CoV-2 infection and interpretation of microbiological results. Clin Microbiol Infect 2020;26(9):1178‑82.
17. Altawalah H. Antibody responses to natural SARS-CoV-2 infection or after COVID-19 vaccination. Vaccines [Internet]. Août 2021 [consulté le 11 février 2022]; Disponible sur : https://doi.org/10.3390/vaccines9080910
18. Direction générale de la santé. Recommandations pour la protection des personnes sévèrement immunodéprimées contre le Covid-19 (vaccination et prophylaxie primaire) – 19 novembre 2021 [Internet]. Disponible sur : https://solidarites-sante.gouv.fr/IMG/pdf/cosv_-_recommandations_pour_la_protection_des_personnes_severement_immunodeprimees_-_19_novembre_2021.pdf
19. Tegally H, Wilkinson E, Giovanetti M, Iranzadeh A, Fonseca V, Giandhari J, et al. Detection of a SARS-CoV-2 variant of concern in South Africa. Nature 2021;592(7854):438‑43.
20. Cele S, Karim F, Lustig G, San JE, Hermanus T, Tegally H, et al. SARS-CoV-2 prolonged infection during advanced HIV disease evolves extensive immune escape. Cell Host Microbe 2022;30(2):154‑62.
21. Contribution du Conseil scientifique COVID-19. Avis du 8 février 2022 - Santé humaine, animale, environnement : les leçons de la crise [Internet]. Disponible sur : https://solidarites-sante.gouv.fr/IMG/pdf/contribution_conseil_scientifique_8_fevrier_2022_one_health.pdf
22. Garry RF. Mutations arising in SARS-CoV-2 spike on sustained human-to-human transmission and human-to-animal passage [Internet]. Virological.org. 2021. Disponible sur : https://virological.org/t/mutations-arising-in-sars-cov-2-spike-on-sustained-human-to-human-transmission-and-human-to-animal-passage/578
23. Organisation mondiale de la santé. Suivi des variants du SARS-CoV-2 - Février 2020 [Internet]. Disponible sur : https://www.who.int/fr/activities/tracking-SARS-CoV-2-variants
24. Cameroni E, Bowen JE, Rosen LE, Saliba C, Zepeda SK, Culap K, et al. Broadly neutralizing antibodies overcome SARS-CoV-2 Omicron antigenic shift. Nature 2021;602:664‑70.
25. Santé publique France. Coronavirus : circulation des variants du SARS-CoV-2 [Internet]. Disponible sur : https://www.santepubliquefrance.fr/dossiers/coronavirus-covid-19/coronavirus-circulation-des-variants-du-sars-cov-2#block-270756
2. Coronaviridae Study Group of the International Committee on Taxonomy of Viruses. The species severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol 2020;5(4):536‑44.
3. Walls AC, Park Y-J, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell 2020;180:281‑92.
4. Jackson CB, Farzan M, Chen B, Choe H. Mechanisms of SARS-CoV-2 entry into cells. Nat Rev Mol Cell Biol 2022;23(1):3‑20.
5. Amoutzias GD, Nikolaidis M, Tryfonopoulou E, Chlichlia K, Markoulatos P, Oliver SG. The remarkable evolutionary plasticity of coronaviruses by mutation and recombination: insights for the COVID-19 pandemic and the future evolutionary paths of SARS-CoV-2. Viruses [Internet]. Janvier 2022 [consulté le 1er février 2022]. Disponible sur : https://doi.org/10.3390/v14010078
6. Meyerowitz EA, Richterman A, Gandhi RT, Sax PE. Transmission of SARS-CoV-2: a review of viral, host, and environmental factors. Ann Intern Med 2021;174(1):69‑79.
7. Burki TK. Omicron variant and booster COVID-19 vaccines. Lancet Respir Med [Internet]. Février 2022 [consulté le 23 février 2022]; Disponible sur : https://doi.org/10.1016/S2213-2600(21)00559-2
8. Bwire GM, Majigo MV, Njiro BJ, Mawazo A. Detection profile of SARS-CoV-2 using RT-PCR in different types of clinical specimens: A systematic review and meta-analysis. J Med Virol 2021;93(2):719‑25.
9. Haute Autorité de santé. Méta-analyse de l’intérêt diagnostique des tests RT-PCR salivaires de détection du SARS- CoV-2 - 10 février 2021 [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2021-02/meta-analyse_rt-pcr_salive_vd.pdf
10. Organisation mondiale de la santé. Détection des antigènes pour le diagnostic de l’infection à SARS-CoV-2 - 6 octobre 2020 [Internet]. Disponible sur : https://apps.who.int/iris/bitstream/handle/10665/347019/WHO-2019-nCoV-Antigen-Detection-2021.1-fre.pdf
11. Haute Autorité de santé. Avis n° 2020.0020/AC/SEAP du 6 mars 2020 : détection du génome du coronavirus SARS-CoV-2 par technique de transcription inverse suivie d’une amplification [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2020-03/ac_rt-pcr_sars_cov2_cd_20200306_visasj_v2_post_cd.pdf
12. Société française de dermatologie. Avis du 25 septembre 2020 de la Société française de microbiologie relatif à l’interprétation de la valeur de Ct (estimation de la charge virale) obtenue en cas de RT-PCR SARS-CoV-2 positive sur les prélèvements cliniques réalisés à des fins diagnostiques ou de dépistage, version 4 [Internet]. Disponible sur: https://www.sfm-microbiologie.org/wp-content/uploads/2021/01/Avis-SFM-valeur-Ct-excrétion-virale-_-Version-def-14012021_V4.pdf
13. Kernéis S, Elie C, Fourgeaud J, Choupeaux L, Mercier Delarue S, Alby M-L, et al. Accuracy of saliva and nasopharyngeal sampling for detection of SARS-CoV-2 in community screening. Eur J Clin Microbiol 2021;40(11):2379‑88.
14. Butler-Laporte G, Lawandi A, Schiller I, Yao M, Dendukuri N, McDonald EG, et al. Comparison of saliva and nasopharyngeal swab nucleic acid amplification testing for detection of SARS-CoV-2: a systematic review and meta-analysis. JAMA Intern Med 2021;181(3):353‑60.
15. Haute Autorité de santé. Avis n° 2021.0007/AC/SEAP du 10 février 2021 : Détection du génome du virus SARS-CoV-2 par technique de transcription inverse suivie d’une amplification (RT-PCR) sur prélèvement salivaire [Internet]. Disponible sur : https://www.has-sante.fr/upload/docs/application/pdf/2021-02/ac_2021_0007_rt-pcr_salivaire_covid-19.pdf
16. Caruana G, Croxatto A, Coste AT, Opota O, Lamoth F, Jaton K, et al. Diagnostic strategies for SARS-CoV-2 infection and interpretation of microbiological results. Clin Microbiol Infect 2020;26(9):1178‑82.
17. Altawalah H. Antibody responses to natural SARS-CoV-2 infection or after COVID-19 vaccination. Vaccines [Internet]. Août 2021 [consulté le 11 février 2022]; Disponible sur : https://doi.org/10.3390/vaccines9080910
18. Direction générale de la santé. Recommandations pour la protection des personnes sévèrement immunodéprimées contre le Covid-19 (vaccination et prophylaxie primaire) – 19 novembre 2021 [Internet]. Disponible sur : https://solidarites-sante.gouv.fr/IMG/pdf/cosv_-_recommandations_pour_la_protection_des_personnes_severement_immunodeprimees_-_19_novembre_2021.pdf
19. Tegally H, Wilkinson E, Giovanetti M, Iranzadeh A, Fonseca V, Giandhari J, et al. Detection of a SARS-CoV-2 variant of concern in South Africa. Nature 2021;592(7854):438‑43.
20. Cele S, Karim F, Lustig G, San JE, Hermanus T, Tegally H, et al. SARS-CoV-2 prolonged infection during advanced HIV disease evolves extensive immune escape. Cell Host Microbe 2022;30(2):154‑62.
21. Contribution du Conseil scientifique COVID-19. Avis du 8 février 2022 - Santé humaine, animale, environnement : les leçons de la crise [Internet]. Disponible sur : https://solidarites-sante.gouv.fr/IMG/pdf/contribution_conseil_scientifique_8_fevrier_2022_one_health.pdf
22. Garry RF. Mutations arising in SARS-CoV-2 spike on sustained human-to-human transmission and human-to-animal passage [Internet]. Virological.org. 2021. Disponible sur : https://virological.org/t/mutations-arising-in-sars-cov-2-spike-on-sustained-human-to-human-transmission-and-human-to-animal-passage/578
23. Organisation mondiale de la santé. Suivi des variants du SARS-CoV-2 - Février 2020 [Internet]. Disponible sur : https://www.who.int/fr/activities/tracking-SARS-CoV-2-variants
24. Cameroni E, Bowen JE, Rosen LE, Saliba C, Zepeda SK, Culap K, et al. Broadly neutralizing antibodies overcome SARS-CoV-2 Omicron antigenic shift. Nature 2021;602:664‑70.
25. Santé publique France. Coronavirus : circulation des variants du SARS-CoV-2 [Internet]. Disponible sur : https://www.santepubliquefrance.fr/dossiers/coronavirus-covid-19/coronavirus-circulation-des-variants-du-sars-cov-2#block-270756