Le prix Nobel de chimie 2020 couronne les travaux d’Emmanuelle Charpentier et de Jennifer Doudna portant sur la correction du génome (genome editing) par les nucléases grâce à la technique CRISPR/Cas9 qu’elles ont mise au point.
La contribution majeure des lauréates permettra un jour peut-être de corriger les mutations responsables des maladies génétiques héritées ou acquises. Mais la technique qu’elles ont développée est d’ores et déjà largement répandue dans les laboratoires de recherche pour la création de modèles cellulaires ou animaux de maladies génétiques ou de cancers.
Dans la boîte à ouvrage du genome editing, on trouvait déjà deux types d’aiguilles à retricoter l’ADN : les protéines à doigt de zinc (zinc finger proteins) et des enzymes de restriction artificielles appelées TALEN, contenant un domaine de liaison à l’ADN (TALE) et un domaine ayant la capacité de cliver la double hélice.
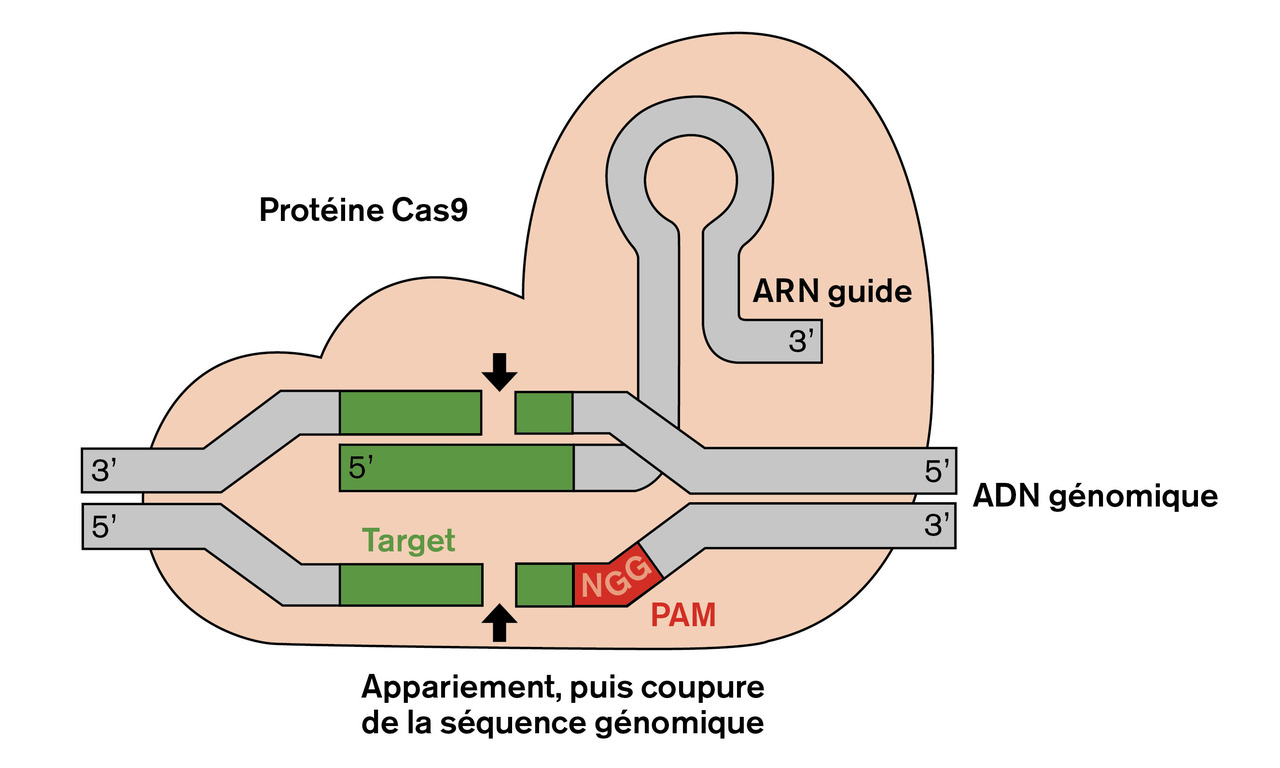
Avec l’outil CRISPR/Cas9, la nucléase Cas9 reconnaît un ARN guide (gRNA) qui permet de cibler une séquence d’ADN complémentaire spécifique. Associée à Cas9, l’ARN guide permet une modification précise de la séquence ciblée par induction de cassures double brin de l’ADN en des sites spécifiques. La séquence d’ADN (protospacer) reconnue par la nucléase Cas9 de Streptococcus pyogenes doit être immédiatement suivie d’une séquence de trois nucléotides (NGG, appelée protospacer adjacent motif [PAM]) pour permettre la liaison de la nucléase Cas9 au double brin d’ADN et sa coupure (v. figure ).
Les cassures double brin de l’ADN ainsi produites sont réparées par deux ordres de mécanismes : la réparation par homologie (homology-directed repair [HDR]) ou la jonction d’extrémités non homologues (non-homologous end joining [NHEJ]). Très fidèle, la réparation par homologie utilise le brin d’ADN complémentaire comme matrice et un oligonucléotide simple brin flanqué de séquences homologues à la séquence cible comme « rustine ». Il est ainsi possible d’obtenir l’intégration site-spécifique d’une séquence d’ADN double brin et la correction (editing) de la mutation. Mais la réparation par homologie ne fonctionne que dans les cellules en division. En l’absence d’activité HDR dans les cellules quiescentes, les cassures des doubles brins d’ADN sont réparées par jonction non homologue (NHEJ), produisant souvent un « KO » de la séquence ciblée au lieu de corriger la mutation.
De ce fait, l’outil CRISPR/Cas9 est surtout utilisé aujourd’hui en laboratoire pour la production de modèles d’inactivation ciblée de gènes par délétions ou insertions de séquences courtes (indels). La nucléase Cas9 peut même conduire à l’apoptose cellulaire par réarrangements chromosomiques étendus (délétions, inversions, translocations). Aussi, parce qu’il expose aux risques de « dommages collatéraux » (off-target), l’outil CRISPR/Cas9 ne peut pas encore être utilisé pour des essais de thérapie génique chez l’homme sans une analyse minutieuse de l’intégrité du génome.
Mais le système est perfectible. Il a été tout récemment montré que l’outil CRISPR pouvait corriger une mutation ponctuelle par substitution de nucléotides (C-base editing, A-base editing) avec peu ou pas de production de cassures double brin. Ce correcteur d’orthographe d’un nouveau genre convertit une paire C-G en T-A ou une paire A-T en G-C. De nouveaux correcteurs sont même capables d’opérer la transversion de paires C-G en G-C. La substitution nucléotidique ponctuelle de la séquence permettrait ainsi la rectification de toute mutation sans cassures indésirables de l’ADN double brin, donc sans risquer les dommages collatéraux et autres modifications « off-target » liées à l’activité de la nucléase Cas9. De surcroît, la rectification nucléotidique ponctuelle serait possible dans les cellules quiescentes en dehors de toute division, par exemple dans les neurones post-mitotiques.
Une autre stratégie encore plus fiable et moins disruptive, liant la nucléase Cas9 partiellement inactivée avec une reverse transcriptase, permettrait de rectifier la séquence, nucléotide par nucléotide, en utilisant un ARN guide (prime editing guide RNA [pegRNA]) comme matrice et permettant de rectifier virtuellement toute anomalie génétique.
Ces avancées spectaculaires font rêver... Elles exposent les professionnels que nous sommes, comme l’opinion, au raccourci saisissant qui consisterait à considérer tout ce qui est techniquement possible au laboratoire comme ipso facto applicable en pratique clinique, éthiquement acceptable et économiquement supportable par notre système de protection sociale, particulièrement en thérapie génique.
Ce serait méconnaître le parcours d’obstacles réglementaire, économique et éthique qui sépare toute innovation de ses applications thérapeutiques en toute sécurité. Les enjeux sanitaires et les attentes de l’opinion sont immenses. Gageons qu’ils ne seront pas déçus.
Les progrès ne sauraient être l’apanage de quelques privilégiés. Le véritable progrès, c’est le partage du progrès.
Dans la boîte à ouvrage du genome editing, on trouvait déjà deux types d’aiguilles à retricoter l’ADN : les protéines à doigt de zinc (zinc finger proteins) et des enzymes de restriction artificielles appelées TALEN, contenant un domaine de liaison à l’ADN (TALE) et un domaine ayant la capacité de cliver la double hélice.
Avec l’outil CRISPR/Cas9, la nucléase Cas9 reconnaît un ARN guide (gRNA) qui permet de cibler une séquence d’ADN complémentaire spécifique. Associée à Cas9, l’ARN guide permet une modification précise de la séquence ciblée par induction de cassures double brin de l’ADN en des sites spécifiques. La séquence d’ADN (protospacer) reconnue par la nucléase Cas9 de Streptococcus pyogenes doit être immédiatement suivie d’une séquence de trois nucléotides (NGG, appelée protospacer adjacent motif [PAM]) pour permettre la liaison de la nucléase Cas9 au double brin d’ADN et sa coupure (
Les cassures double brin de l’ADN ainsi produites sont réparées par deux ordres de mécanismes : la réparation par homologie (homology-directed repair [HDR]) ou la jonction d’extrémités non homologues (non-homologous end joining [NHEJ]). Très fidèle, la réparation par homologie utilise le brin d’ADN complémentaire comme matrice et un oligonucléotide simple brin flanqué de séquences homologues à la séquence cible comme « rustine ». Il est ainsi possible d’obtenir l’intégration site-spécifique d’une séquence d’ADN double brin et la correction (editing) de la mutation. Mais la réparation par homologie ne fonctionne que dans les cellules en division. En l’absence d’activité HDR dans les cellules quiescentes, les cassures des doubles brins d’ADN sont réparées par jonction non homologue (NHEJ), produisant souvent un « KO » de la séquence ciblée au lieu de corriger la mutation.
De ce fait, l’outil CRISPR/Cas9 est surtout utilisé aujourd’hui en laboratoire pour la production de modèles d’inactivation ciblée de gènes par délétions ou insertions de séquences courtes (indels). La nucléase Cas9 peut même conduire à l’apoptose cellulaire par réarrangements chromosomiques étendus (délétions, inversions, translocations). Aussi, parce qu’il expose aux risques de « dommages collatéraux » (off-target), l’outil CRISPR/Cas9 ne peut pas encore être utilisé pour des essais de thérapie génique chez l’homme sans une analyse minutieuse de l’intégrité du génome.
Mais le système est perfectible. Il a été tout récemment montré que l’outil CRISPR pouvait corriger une mutation ponctuelle par substitution de nucléotides (C-base editing, A-base editing) avec peu ou pas de production de cassures double brin. Ce correcteur d’orthographe d’un nouveau genre convertit une paire C-G en T-A ou une paire A-T en G-C. De nouveaux correcteurs sont même capables d’opérer la transversion de paires C-G en G-C. La substitution nucléotidique ponctuelle de la séquence permettrait ainsi la rectification de toute mutation sans cassures indésirables de l’ADN double brin, donc sans risquer les dommages collatéraux et autres modifications « off-target » liées à l’activité de la nucléase Cas9. De surcroît, la rectification nucléotidique ponctuelle serait possible dans les cellules quiescentes en dehors de toute division, par exemple dans les neurones post-mitotiques.
Une autre stratégie encore plus fiable et moins disruptive, liant la nucléase Cas9 partiellement inactivée avec une reverse transcriptase, permettrait de rectifier la séquence, nucléotide par nucléotide, en utilisant un ARN guide (prime editing guide RNA [pegRNA]) comme matrice et permettant de rectifier virtuellement toute anomalie génétique.
Ces avancées spectaculaires font rêver... Elles exposent les professionnels que nous sommes, comme l’opinion, au raccourci saisissant qui consisterait à considérer tout ce qui est techniquement possible au laboratoire comme ipso facto applicable en pratique clinique, éthiquement acceptable et économiquement supportable par notre système de protection sociale, particulièrement en thérapie génique.
Ce serait méconnaître le parcours d’obstacles réglementaire, économique et éthique qui sépare toute innovation de ses applications thérapeutiques en toute sécurité. Les enjeux sanitaires et les attentes de l’opinion sont immenses. Gageons qu’ils ne seront pas déçus.
Les progrès ne sauraient être l’apanage de quelques privilégiés. Le véritable progrès, c’est le partage du progrès.