La métagénomique clinique, nouvel outil de biologie moléculaire, permet de détecter l’ensemble des micro-organismes présents dans tous types de prélèvements. Son utilisation est plus ou moins pertinente selon le type d’infection : infections du système nerveux central, respiratoires, digestives, oculaires, cutanées... Des études supplémentaires sont nécessaires pour déterminer sa place dans l’arsenal diagnostique en infectiologie.
Les méthodes conventionnelles de microbiologie incluent les examens macroscopiques et microscopiques, la culture pour les micro-organismes cultivables, les sérologies, les PCR (polymerase chain reaction) spécifiques ou multiplexes syndromiques, et le séquençage des zones conservées de bactéries (16S) et des champignons (ITS). Ces méthodes ne permettent pas toujours l’identification d’un micro-organisme lors d’une infection. Actuellement, de nombreuses infections restent sans cause identifiée : par exemple, 50 % des méningites et encéphalites.1 En effet, l’utilisation des méthodes conventionnelles peut être limitée par une faible charge microbienne initiale dans le prélèvement, l’administration précoce d’un traitement probabiliste, ou un délai de pousse prolongé.
La métagénomique clinique (MgC) est un nouvel outil diagnostique dans l’arsenal des microbiologistes. Alors que la majorité des tests de biologie moléculaire est spécifique d’un ou plusieurs micro-organismes, la MgC permet de rechercher tous les acides nucléiques présents dans un échantillon et d’identifier ainsi potentiellement tous les micro-organismes.
La MgC peut également permettre de suivre l’évolution génétique du micro-organisme, de déterminer sa phylogénie ou de prédire sa résistance au traitement.2
La métagénomique clinique (MgC) est un nouvel outil diagnostique dans l’arsenal des microbiologistes. Alors que la majorité des tests de biologie moléculaire est spécifique d’un ou plusieurs micro-organismes, la MgC permet de rechercher tous les acides nucléiques présents dans un échantillon et d’identifier ainsi potentiellement tous les micro-organismes.
La MgC peut également permettre de suivre l’évolution génétique du micro-organisme, de déterminer sa phylogénie ou de prédire sa résistance au traitement.2
Mode d’emploi de la métagénomique clinique
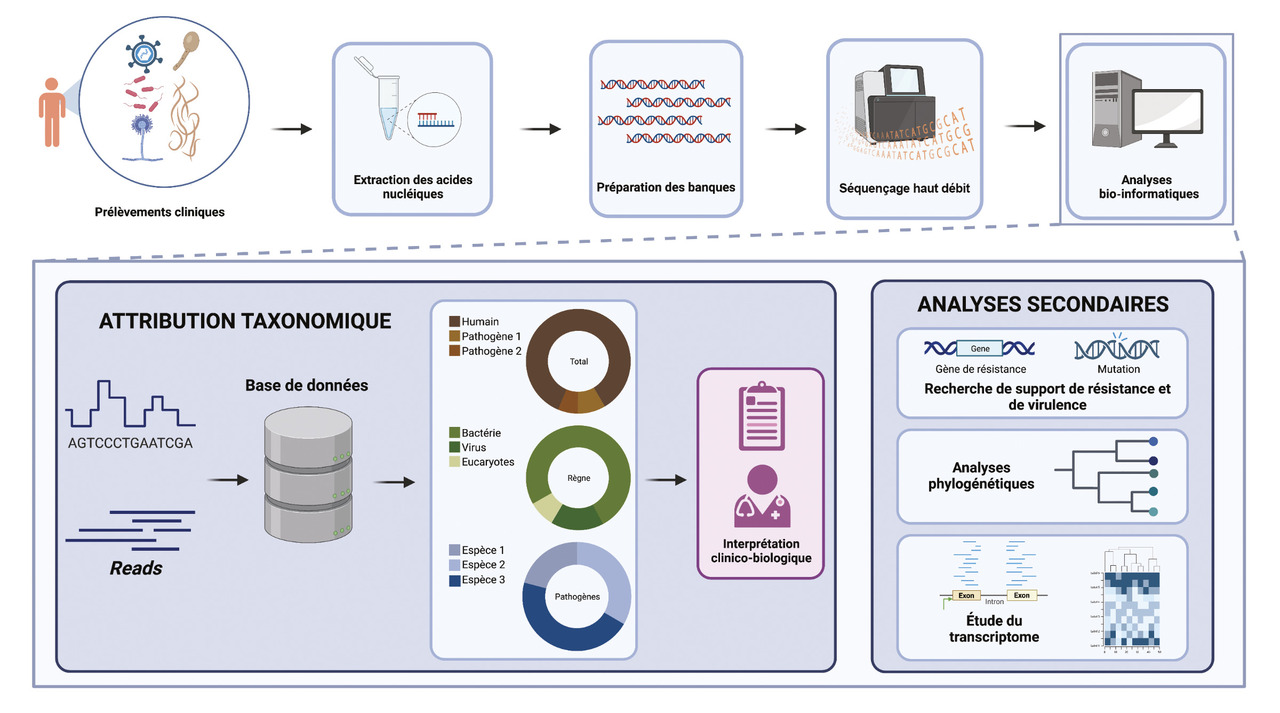
La MgC peut être réalisée sur tout type d’échantillons cliniques (sang, urine, prélèvements respiratoires, prélèvements profonds, liquide céphalorachidien [LCR], biopsies).
Analyses biologiques
Extraction des acides nucléiques et préparation des banques
La première étape consiste à extraire tous les acides nucléiques (ADN et ARN) présents dans l’échantillon. Après l’extraction, des banques de séquençage sont construites, consistant en la fragmentation en petites séquences d’ADN, l’ajout d’adaptateur et d’index, une amplification et une purification des acides nucléiques. Les banques sont ensuite chargées sur une plateforme de séquençage.Séquençage
La MgC repose sur les nouvelles technologies de next generation sequencing (NGS), ou séquençage haut débit, permettant de générer un très grand nombre de séquences. La méthode la plus connue est le séquençage par amplification en pont (Illumina), générant des séquences courtes (environ 300 paires de bases [pb]). Récemment, de nouvelles méthodes de séquençage dites de troisième génération (Pacific Biosciences et Nanopore) sont apparues, perrmettant de séquencer des séquences plus longues (plusieurs dizaines de kilobases).En MgC, l’utilisation des analyses non ciblées de type shotgun sont privilégiées. Elles ne nécessitent pas d’amplification ciblée et permettent de séquencer la totalité de l’ADN et/ou de l’ARN, et peuvent ainsi identifier tous types de micro-organismes (bactéries, virus, champignons, parasites) en un seul test. Malgré des quantités élevées de micro-organismes dans certains compartiments comme le tube digestif, la taille du génome humain est 100 à 106 fois plus grande que celle des génomes microbiens et il est donc présent en quantité plus importante. Il constitue la majorité de l’ADN et de l’ARN séquencé. Les acides nucléiques des micro-organismes représentent en général moins de 1 % de la totalité des acides nucléiques présents dans l’échantillon.
Analyses bio-informatiques
Les technologies de NGS génèrent un nombre de données important, demandant des capacités de stockage et de traitement conséquentes. Les séquences obtenues après séquençage sont appelées reads. Compte tenu de la relativement faible quantité d’acides nucléiques microbiens présents dans les échantillons cliniques, la documentation microbiologique nécessite un nombre important de reads. Après avoir éliminé les reads ne respectant pas les critères de taille et de qualité de séquençage, ainsi que ceux apparentés au génome humain, les reads restants sont comparés aux séquences contenues dans des bases de données taxonomiques bactériennes, virales, parasitaires et fongiques afin d’identifier les micro-organismes présents dans l’échantillon. De plus, peuvent être réalisées des analyses phylogénétiques, de recherche de supports de résistance et de virulence ainsi que des analyses transcriptomiques microbiennes (fig. 1 ).
Apports de la métagénomique en infectiologie
La MgC est plus ou moins pertinente selon les localisations infectieuses, et des études complémentaires sont nécessaires.
Infections du système nerveux central
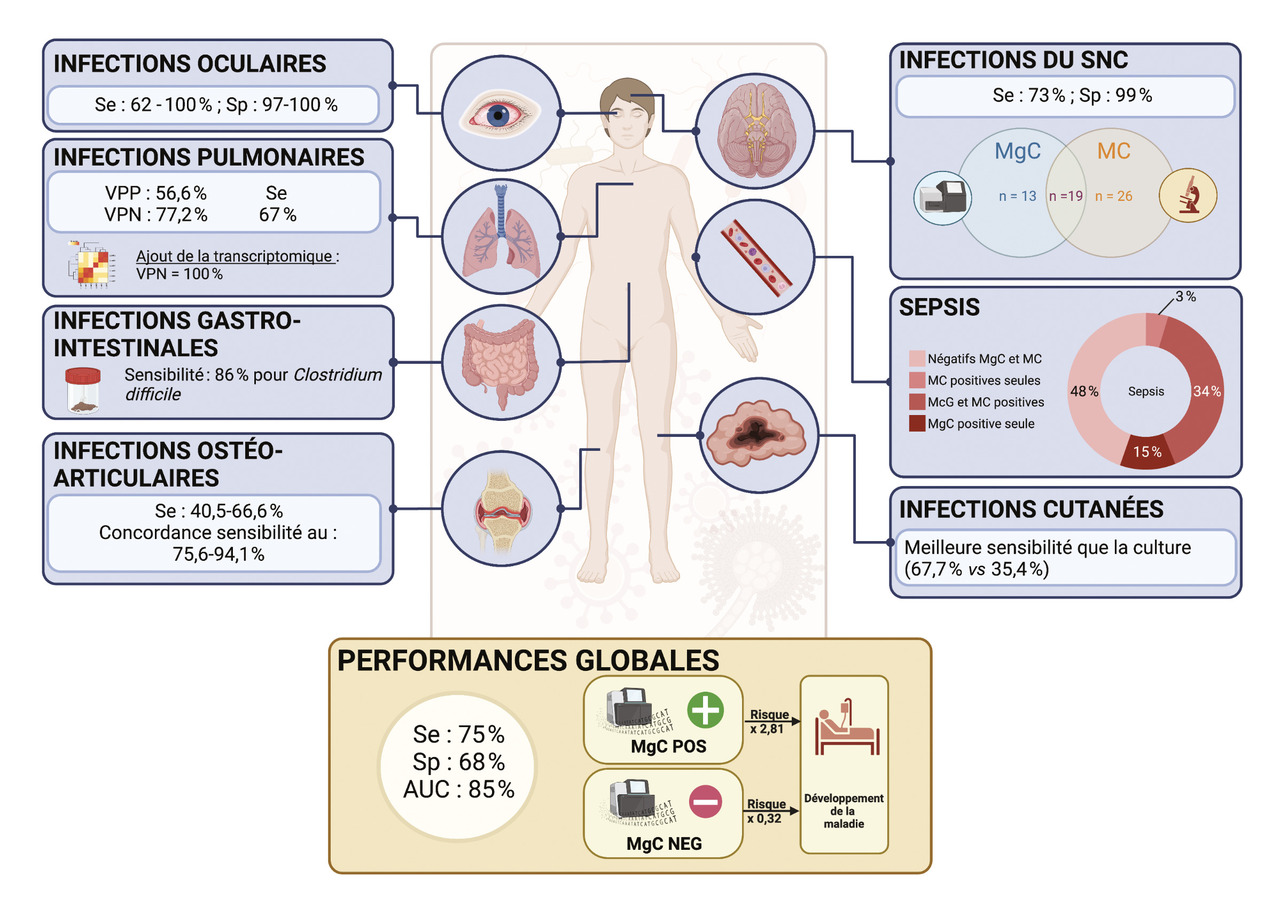
Dans près de la moitié des cas de méningites et d’encéphalites, aucun micro-organisme n’est identifié.1 La sensibilité et la spécificité de la MgC sur le liquide céphalo-rachidien (LCR) ont été estimées respectivement à 73 % et 99 %.1 La première étude prospective de grande ampleur de l’apport de la MgC a été réalisée sur 204 LCR prélevés dans un contexte de suspicion de méningite, d’encéphalite ou de myélite. Cinquante-huit infections ont été diagnostiquées chez 57 patients par MgC ou méthodes microbiologiques conventionnelles. Parmi elles, 22 % (n = 13) ont été diagnostiquées seulement par MgC, 33 % (n = 19) par MgC et méthodes conventionnelles et 45 % (n = 26) par méthodes conventionnelles uniquement (11 diagnostics réalisés par sérologie, 7 réalisés sur un autre prélèvement et 8 négatifs en raison de faibles concentrations en micro-organisme dans le LCR). Concernant les 13 infections diagnostiquées seulement par MgC, le résultat de MgC a eu un impact dans la prise en charge clinique pour 8 d’entre elles, avec un changement de traitement pour 7 d’entre elles.1 D’autres études prospectives sont nécessaires afin de déterminer la place de la MgC dans l’algorithme diagnostique des infections du système nerveux central.
Infections respiratoires
Les infections des voies respiratoires sont une cause majeure de mortalité et de morbidité, et 30 à 60 % des cas de pneumonies communautaires restent sans cause identifiée. L’utilisation de la MgC dans le cas d’infections respiratoires basses a été évaluée dans une étude prospective incluant 92 patients avec syndrome de détresse respiratoire aiguë (SDRA).3 En une seule analyse, la MgC a pu identifier les 38 micro-organismes retrouvés par les méthodes conventionnelles chez les 26 patients infectés, mais elle a également permis d’étudier la flore microbienne respiratoire et de la comparer à celle des patients non infectés, mettant ainsi en exergue une diversité microbienne moindre chez les patients infectés. Enfin, en combinant, grâce à un algorithme, les données sur les micro-organismes détectés, le microbiome et le transcriptome, une valeur prédictive négative (VPN) de 100 % a pu être établie pour la MgC. Cette méthode semble donc prometteuse dans les SDRA. L’intérêt de son utilisation dans d’autres infections du système respiratoire, souvent complexes, graves et de causes diverses, reste encore néanmoins à démontrer.
Infections de la peau et des tissus mous
Peu d’études utilisent la MgC dans les cas d’infections de la peau et des tissus mous. Dans une étude rétrospective réalisée sur 96 patients, la MgC a montré une meilleure sensibilité que la culture bactérienne (67,7 % versus 35,4 %), détectant également les virus et les bactéries anaérobies en un seul test.4 De plus, la proportion de prélèvements polymicrobiens détectés par MgC était plus élevée que celle détectée par culture. L’apport de la MgC dans les infections cutanées a été recensé sur des cohortes de petite taille, notamment sur 34 cas de fasciites nécrosantes5 ou sur 98 patients atteints d’exanthèmes aigus.6 Cependant, l’interprétation des résultats peut être complexe du fait de la présence du microbiome cutané.
Infections oculaires
Les infections oculaires peuvent atteindre plusieurs zones anatomiques de l’œil et sont causées par divers micro-organismes. Les analyses sont souvent limitées en raison de la faible quantité d’échantillon disponible par rapport à un grand nombre de causes infectieuses possible, rendant le diagnostic difficile. La preuve de concept de l’utilisation de la MgC pour les infections oculaires a été démontrée par Doan et al.7 en testant trois humeurs aqueuses connues positives à Cryptococcus neoformans, Toxoplasma gondii, et Herpes simplex virus de type 1. De plus, le virus de la rubéole a été retrouvé dans une quatrième humeur aqueuse chez un patient atteint d’uvéite bilatérale idiopathique datant de plusieurs années. Les performances de la MgC pour les infections oculaires ont été évaluées chez 46 patients atteints de kératites. Selon le micro-organisme détecté (bactérie ou champignon) et la matrice utilisée en MgC (ADN ou ARN), la sensibilité varie entre 62 et 100 % et la spécificité entre 97 et 100 %.8 Enfin, dans une étude rétrospective sur 14 patients atteints de conjonctivite,9 la MgC a pu identifier de l’adénovirus D8 dans 86 % des cas (n = 12) et Vittaforma cornea (phylum des microsporidies) dans 14 % (n = 2). Des analyses complémentaires de transcriptomique ont également pu mettre en évidence une réponse cellulaire différente selon le micro-organisme détecté. Ces analyses pourraient permettre de détecter ces micro-organismes même lorsque la charge microbienne est trop faible pour une identification directe.
Infections du système digestif
La métagénomique ciblée est largement utilisée dans l’étude du microbiome digestif. Cependant, peu d’études s’intéressent à l’apport de la MgC dans le diagnostic des infections digestives. Dans une étude rétrospective portant sur 22 selles diarrhéiques, une sensibilité de 86 % a été obtenue en MgC pour la recherche de Clostridioides difficile. Elle a également permis la détection simultanée de C. perfringens (1/22), et de Norovirus, de Sapovirus, d’Anellovirus et de Parechovirus dans 27,3 % (n = 6) des selles.10 La MgC a également été utilisée pour analyser des prélèvements de selles connues positives pour des infections parasitaires par méthodes conventionnelles (microscopie optique couplée à la PCR), retrouvant des résultats concordants.11
Infections ostéoarticulaires
Les infections ostéoarticulaires sont des infections graves qui nécessitent un traitement antibiotique adapté et prolongé. Pourtant, le diagnostic par méthodes conventionnelles – essentiellement et permettent sur la culture – manque de sensibilité, en particulier pour les bactéries difficilement cultivables. La MgC pourrait donc apparaître comme complémentaire aux méthodes conventionnelles.
D’Humières et al.12 ont comparé les performances de la MgC versus la culture bactérienne et le séquençage de l’ARN 16S lors d’infections ostéoarticulaires. Pour 33 des 34 patients, la MgC a soit confirmé le diagnostic (17/34), soit complété le diagnostic établi (17/34). La MgC a permis d’identifier 72 bactéries non retrouvées en culture, principalement des bactéries anaérobies. Néanmoins, la sensibilité est de 66,6 % pour les infections monomicrobiennes, et de 40,5 % pour les infections polymicrobiennes.
D’Humières et al.12 ont comparé les performances de la MgC versus la culture bactérienne et le séquençage de l’ARN 16S lors d’infections ostéoarticulaires. Pour 33 des 34 patients, la MgC a soit confirmé le diagnostic (17/34), soit complété le diagnostic établi (17/34). La MgC a permis d’identifier 72 bactéries non retrouvées en culture, principalement des bactéries anaérobies. Néanmoins, la sensibilité est de 66,6 % pour les infections monomicrobiennes, et de 40,5 % pour les infections polymicrobiennes.
Sepsis et détection de micro-organismes dans le sang
L’ADN libre (cell-free DNA [cfDNA]) correspond à l’ADN présent en dehors du noyau cellulaire et pouvant être détecté dans les fluides biologiques comme le sang. La présence de cfDNA microbien dans le sang permet de détecter non seulement les micro-organismes lors d’infections disséminées mais également les micro-organismes de certaines infections localisées. Blauwkamp et al.13 ont évalué un test de détection de cfDNA (test Karius) dans le sang chez des patients présentant un sepsis. Un taux de positivité supérieur a pu être obtenu par MgC par rapport aux hémocultures initiales (169/348 versus 63/348) ou par rapport aux autres tests microbiologiques effectués (132/348). De plus, pour les patients ayant reçu une antibiothérapie avant les prélèvements, la MgC avait également un meilleur taux de détection des micro-organismes que les hémocultures (46/94 vs 19/96). Malgré de bonnes performances analytiques, le bénéfice de la MgC dans le sang, en clinique, par rapport aux méthodes conventionnelles semble, pour le moment, limité à une population de patients immunodéprimés.
Performances globales de la MgC
Dans une méta-analyse,14 la sensibilité totale de la MgC, toutes infections confondues, a été évaluée à 75 % et la spécificité à 68 %. De plus, il a été montré que le fait d’avoir un test de MgC positif entraînait un risque 2,81 fois supérieur de développer la maladie. A contrario, le risque de développer la maladie est réduit lorsque les résultats de MgC sont négatifs (fig. 2 ).
Métagénomique clinique : au-delà du diagnostic infectieux
Recherche de supports de résistance
La MgC permet non seulement d’établir un diagnostic microbiologique mais également de rechercher des mutations ou d’étudier l’expression de gènes de résistance et de virulence à partir des données séquencées. Contrairement aux analyses par PCR ciblées, il est possible d’obtenir toutes les séquences des gènes de résistance d’intérêt en un seul test.15 Cependant, certains gènes de résistance peuvent être présents chez les bactéries pathogènes comme chez les commensales, rendant l’implication phénotypique difficile.16 En virologie, la MgC donne potentiellement accès au génome complet du virus et permet donc d’identifier les substitutions associées à la résistance aux antiviraux.2
Étude du transcriptome
Tout comme la recherche de résistance, l’étude du transcriptome peut être réalisée de manière concomitante à la recherche de micro-organismes. La transcriptomique peut être utilisée pour étudier la réponse de l’hôte lors d’une infection. Langelier et al.3 ont mis en évidence sur les infections respiratoires basses une différence d’expression génique par les cellules de l’hôte entre les patients infectés et les non infectés. Il est également possible de déterminer si la cause infectieuse est d’origine bactérienne ou virale grâce à la surexpression de certains gènes spécifiques en fonction de la cause. L’utilisation des données issues de l’analyse du transcriptome offre de nouvelles pistes importantes dans le diagnostic.
Épidémiologie
Lors des épidémies, l’identification des cas suspects est essentielle pour mettre en place les mesures sanitaires nécessaires. L’un des exemples emblématiques récents de l’utilisation de la technologie de MgC en épidémiologie a été celui de la pandémie de Covid-19. En effet, la MgC a permis d’identifier et de décrire pour la première fois le SARS-CoV-2,17 de suivre son évolution au cours de temps, détectant ainsi l’apparition de nouveaux variants.
Avantages et limites de la métagénomique clinique
La MgC offre la possibilité de détecter sans a priori des micro-organismes de nature différente en un seul test de manière exhaustive. Cette technologie permet également la détection de micro-organismes inhabituels, de micro-organismes nouveaux ou avec une présentation clinique atypique.1 L’implication des germes retrouvés dans la pathologie peut s’avérer peu claire.
Des réunions de concertation multidisciplinaire associant microbiologistes, cliniciens, infectiologues peuvent avoir lieu afin d’établir le bon diagnostic pour optimiser la prise en charge.
La MgC est, à l’heure actuelle, réalisée seulement dans certains centres hospitalo-universitaires (CHU). Le délai de réalisation et de rendu des résultats est, en routine, d’une à deux semaines. De plus, une des principales limites de la MgC est son coût : les équipements et les réactifs sont onéreux. L’analyse est cotée RIHN942, donc facturée 2 205,90 euros, mais le coût réel pour le laboratoire varie selon les séquenceurs disponibles et la quantité d’analyses réalisées.
Son implémentation nécessite une infrastructure dédiée avec une expertise en bio-informatique. Cependant, la mise sur le marché de séquenceurs moins chers ainsi que le développement de tests commerciaux permettront l’utilisation de la MgC dans un plus grand nombre de laboratoires dans les années futures.
Des réunions de concertation multidisciplinaire associant microbiologistes, cliniciens, infectiologues peuvent avoir lieu afin d’établir le bon diagnostic pour optimiser la prise en charge.
La MgC est, à l’heure actuelle, réalisée seulement dans certains centres hospitalo-universitaires (CHU). Le délai de réalisation et de rendu des résultats est, en routine, d’une à deux semaines. De plus, une des principales limites de la MgC est son coût : les équipements et les réactifs sont onéreux. L’analyse est cotée RIHN942, donc facturée 2 205,90 euros, mais le coût réel pour le laboratoire varie selon les séquenceurs disponibles et la quantité d’analyses réalisées.
Son implémentation nécessite une infrastructure dédiée avec une expertise en bio-informatique. Cependant, la mise sur le marché de séquenceurs moins chers ainsi que le développement de tests commerciaux permettront l’utilisation de la MgC dans un plus grand nombre de laboratoires dans les années futures.
Des perspectives prometteuses en infectiologie
La MgC apparaît comme un outil prometteur en infectiologie. La diminution des coûts de NGS et l’amélioration des capacités de calcul et de stockage la rendent accessible à un plus grand nombre de laboratoires. Cette technique offre aux microbiologistes la possibilité de relever de nouveaux défis tels que l’identification exhaustive de micro-organismes nouveaux, difficilement cultivables ou décapités par un traitement préalable. Cependant, malgré les progrès visant à réduire les coûts, la MgC reste, encore à l’heure actuelle, plus onéreuse que les méthodes conventionnelles. Sa place dans l’arsenal diagnostique en infectiologie reste à préciser lors de larges études prospectives.
Références
1. Wilson MR, Sample HA, Zorn KC, Arevalo S, Yu G, Neuhaus J, et al. Clinical metagenomic sequencing for diagnosis of meningitis and encephalitis. N Engl J Med 2019;380(24):2327-40.
2. d’Humières C, Salmona M, Dellière S, Leo S, Rodriguez C, Angebault C, et al. The potential role of clinical metagenomics in infectious diseases: Therapeutic perspectives. Drugs 2021;81(13):1453-66.
3. Langelier C, Kalantar KL, Moazed F, Wilson MR, Crawford ED, Deiss T, et al. Integrating host response and unbiased microbe detection for lower respiratory tract infection diagnosis in critically ill adults. Proc Natl Acad Sci USA 2018;115(52):E12353-62.
4. Wang Q, Miao Q, Pan J, Jin W, Ma Y, Zhang Y, et al. The clinical value of metagenomic next-generation sequencing in the microbiological diagnosis of skin and soft tissue infections. Int J Infect Dis 2020;100:414-20.
5. Rodriguez C, Jary A, Hua C, Woerther PL, Bosc R, Desroches M, et al. Pathogen identification by shotgun metagenomics of patients with necrotizing soft-tissue infections. Br J Dermatol 2020;183(1):105-13.
6. Deschamps O, Ortonne N, Hüe S, Rodriguez C, Deschodt C, Hirsch G, et al. Acute exanthemas: A prospective study of 98 adult patients with an emphasis on cytokinic and metagenomic investigation. Br J Dermatol 2020;182(2):355-63.
7. Doan T, Wilson MR, Crawford ED, Chow ED, Khan LM, Knopp KA, et al. Illuminating uveitis: Metagenomic deep sequencing identifies common and rare pathogens. Genome Med 2016;8(1):90.
8. Lalitha P, Prajna NV, Sikha M, Gunasekaran R, Hinterwirth A, Worden L, et al. Evaluation of metagenomic deep sequencing as a diagnostic test for infectious keratitis. Ophthalmology 2021;128(3):473-5.
9. Lalitha P, Seitzman GD, Kotecha R, Hinterwirth A, Chen C, Zhong L, et al. Unbiased pathogen detection and host gene profiling for conjunctivitis. Ophthalmology 2019;126(8):1090-4.
10. Zhou Y, Wylie KM, El Feghaly RE, Mihindukulasuriva KA, Elward A, Haslam DB, et al. Metagenomic approach for identification of the pathogens associated with diarrhea in stool specimens. J Clin Microbiol 2016;54(2):368-75.
11. Wylezich C, Caccio SM, Walochnik J, Beer M, Höper D. Untargeted metagenomics shows a reliable performance for synchronous detection of parasites. Parasitol Res 2020;119(8):2623-9.
12. d’Humières C, Gaïa N, Gueye S, de Lastours V, Leflon-Guibout V, Maataoui N, et al. Contribution of clinical metagenomics to the diagnosis of bone and joint infections. Front Microbiol 2022;13:863777.
13. Blauwkamp TA, Thair S, Rosen MJ, Blair L, Lindner MS, Vilfan ID, et al. Analytical and clinical validation of a microbial cell-free DNA sequencing test for infectious disease. Nat Microbiol 2019;4(4):663-74.
14. Liu J, Zhang Q, Dong YQ, Yin J, Qiu YQ. Diagnostic accuracy of metagenomic next-generation sequencing in diagnosing infectious diseases: A meta-analysis. Sci Rep 2022;12(1):21032.
15. Serpa PH, Deng X, Abdelghany M, Crawford E, Malcolm K, Caldera S, et al. Metagenomic prediction of antimicrobial resistance in critically ill patients with lower respiratory tract infections. Genome Med 2022;14(1):74.
16. Ruppé E, Ghozlane A, Tap J, Pons N, Alvarez AS, Maziers N, et al. Prediction of the intestinal resistome by a three-dimensional structure-based method. Nat Microbiol 2019;4(1):112-23.
17. Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, et al. A new coronavirus associated with human respiratory disease in China. Nature 2020;579(7798):265-9.
2. d’Humières C, Salmona M, Dellière S, Leo S, Rodriguez C, Angebault C, et al. The potential role of clinical metagenomics in infectious diseases: Therapeutic perspectives. Drugs 2021;81(13):1453-66.
3. Langelier C, Kalantar KL, Moazed F, Wilson MR, Crawford ED, Deiss T, et al. Integrating host response and unbiased microbe detection for lower respiratory tract infection diagnosis in critically ill adults. Proc Natl Acad Sci USA 2018;115(52):E12353-62.
4. Wang Q, Miao Q, Pan J, Jin W, Ma Y, Zhang Y, et al. The clinical value of metagenomic next-generation sequencing in the microbiological diagnosis of skin and soft tissue infections. Int J Infect Dis 2020;100:414-20.
5. Rodriguez C, Jary A, Hua C, Woerther PL, Bosc R, Desroches M, et al. Pathogen identification by shotgun metagenomics of patients with necrotizing soft-tissue infections. Br J Dermatol 2020;183(1):105-13.
6. Deschamps O, Ortonne N, Hüe S, Rodriguez C, Deschodt C, Hirsch G, et al. Acute exanthemas: A prospective study of 98 adult patients with an emphasis on cytokinic and metagenomic investigation. Br J Dermatol 2020;182(2):355-63.
7. Doan T, Wilson MR, Crawford ED, Chow ED, Khan LM, Knopp KA, et al. Illuminating uveitis: Metagenomic deep sequencing identifies common and rare pathogens. Genome Med 2016;8(1):90.
8. Lalitha P, Prajna NV, Sikha M, Gunasekaran R, Hinterwirth A, Worden L, et al. Evaluation of metagenomic deep sequencing as a diagnostic test for infectious keratitis. Ophthalmology 2021;128(3):473-5.
9. Lalitha P, Seitzman GD, Kotecha R, Hinterwirth A, Chen C, Zhong L, et al. Unbiased pathogen detection and host gene profiling for conjunctivitis. Ophthalmology 2019;126(8):1090-4.
10. Zhou Y, Wylie KM, El Feghaly RE, Mihindukulasuriva KA, Elward A, Haslam DB, et al. Metagenomic approach for identification of the pathogens associated with diarrhea in stool specimens. J Clin Microbiol 2016;54(2):368-75.
11. Wylezich C, Caccio SM, Walochnik J, Beer M, Höper D. Untargeted metagenomics shows a reliable performance for synchronous detection of parasites. Parasitol Res 2020;119(8):2623-9.
12. d’Humières C, Gaïa N, Gueye S, de Lastours V, Leflon-Guibout V, Maataoui N, et al. Contribution of clinical metagenomics to the diagnosis of bone and joint infections. Front Microbiol 2022;13:863777.
13. Blauwkamp TA, Thair S, Rosen MJ, Blair L, Lindner MS, Vilfan ID, et al. Analytical and clinical validation of a microbial cell-free DNA sequencing test for infectious disease. Nat Microbiol 2019;4(4):663-74.
14. Liu J, Zhang Q, Dong YQ, Yin J, Qiu YQ. Diagnostic accuracy of metagenomic next-generation sequencing in diagnosing infectious diseases: A meta-analysis. Sci Rep 2022;12(1):21032.
15. Serpa PH, Deng X, Abdelghany M, Crawford E, Malcolm K, Caldera S, et al. Metagenomic prediction of antimicrobial resistance in critically ill patients with lower respiratory tract infections. Genome Med 2022;14(1):74.
16. Ruppé E, Ghozlane A, Tap J, Pons N, Alvarez AS, Maziers N, et al. Prediction of the intestinal resistome by a three-dimensional structure-based method. Nat Microbiol 2019;4(1):112-23.
17. Wu F, Zhao S, Yu B, Chen YM, Wang W, Song ZG, et al. A new coronavirus associated with human respiratory disease in China. Nature 2020;579(7798):265-9.