Comment justifier a posteriori un accident toxique médicamenteux s’il existe des moyens de prévenir sa survenue ?
L’étude du génome humain a permis d’établir le lien entre les variations de séquence de l’ADN et la susceptibilité individuelle aux maladies mais également à l’exposition à des composés chimiques environnementaux désignés sous le terme général de xénobiotiques. De façon inattendue, l’analyse comparative des gènes de l’homme de Néandertal et de l’Homo sapiens actuel a mis en évidence l’origine ancestrale de ce lien. Ainsi les populations caucasiennes, ayant hérité d’une part plus importante du génome néander- talien par comparaison avec d’autres populations, sont plus sensibles à la toxicité de certains xénobiotiques.1 Dans un contexte thérapeutique, la pharmacogénétique étudie les mécanismes d’origine génétique intervenant dans la variabilité interindividuelle de la réponse aux médicaments. Elle vise à identifier les individus répondeurs et les non-répondeurs à un médicament donné, à dépister les individus à risque de survenue d’un événement indésirable et, enfin, à prévoir la dose la plus adaptée à chaque patient.
Un bref historique de la pharmacogénétique permet de rappeler que le philosophe grec Pythagore a rapporté au ive siècle avant J.-C. des cas d’intolérance aux fèves associés à des crises hémolytiques aiguës et qu’il faudra attendre la fin du xixe siècle pour que leur caractère héréditaire soit définitivement admis. La démonstration entre le déficit génétique en glucose-6-phosphate déshydrogénase (G6PD) et le favisme est publiée en 1956 dans la revue Blood par W. H. Crosby. Ce même déficit explique également la survenue de crises hémolytiques après la prise de primaquine (antipaludéen) et représente l’un des premiers exemples célèbres illustrant l’existence d’une prédispo-sition génétique aux effets indésirables des médicaments. Enfin, le terme de pharmacogénétique est utilisé pour la première fois en 1959 dans un journal allemand de médecine interne par Friedrich Vogel, qui y décrit le caractère héréditaire de réponses anormales aux médicaments.
Un bref historique de la pharmacogénétique permet de rappeler que le philosophe grec Pythagore a rapporté au ive siècle avant J.-C. des cas d’intolérance aux fèves associés à des crises hémolytiques aiguës et qu’il faudra attendre la fin du xixe siècle pour que leur caractère héréditaire soit définitivement admis. La démonstration entre le déficit génétique en glucose-6-phosphate déshydrogénase (G6PD) et le favisme est publiée en 1956 dans la revue Blood par W. H. Crosby. Ce même déficit explique également la survenue de crises hémolytiques après la prise de primaquine (antipaludéen) et représente l’un des premiers exemples célèbres illustrant l’existence d’une prédispo-sition génétique aux effets indésirables des médicaments. Enfin, le terme de pharmacogénétique est utilisé pour la première fois en 1959 dans un journal allemand de médecine interne par Friedrich Vogel, qui y décrit le caractère héréditaire de réponses anormales aux médicaments.
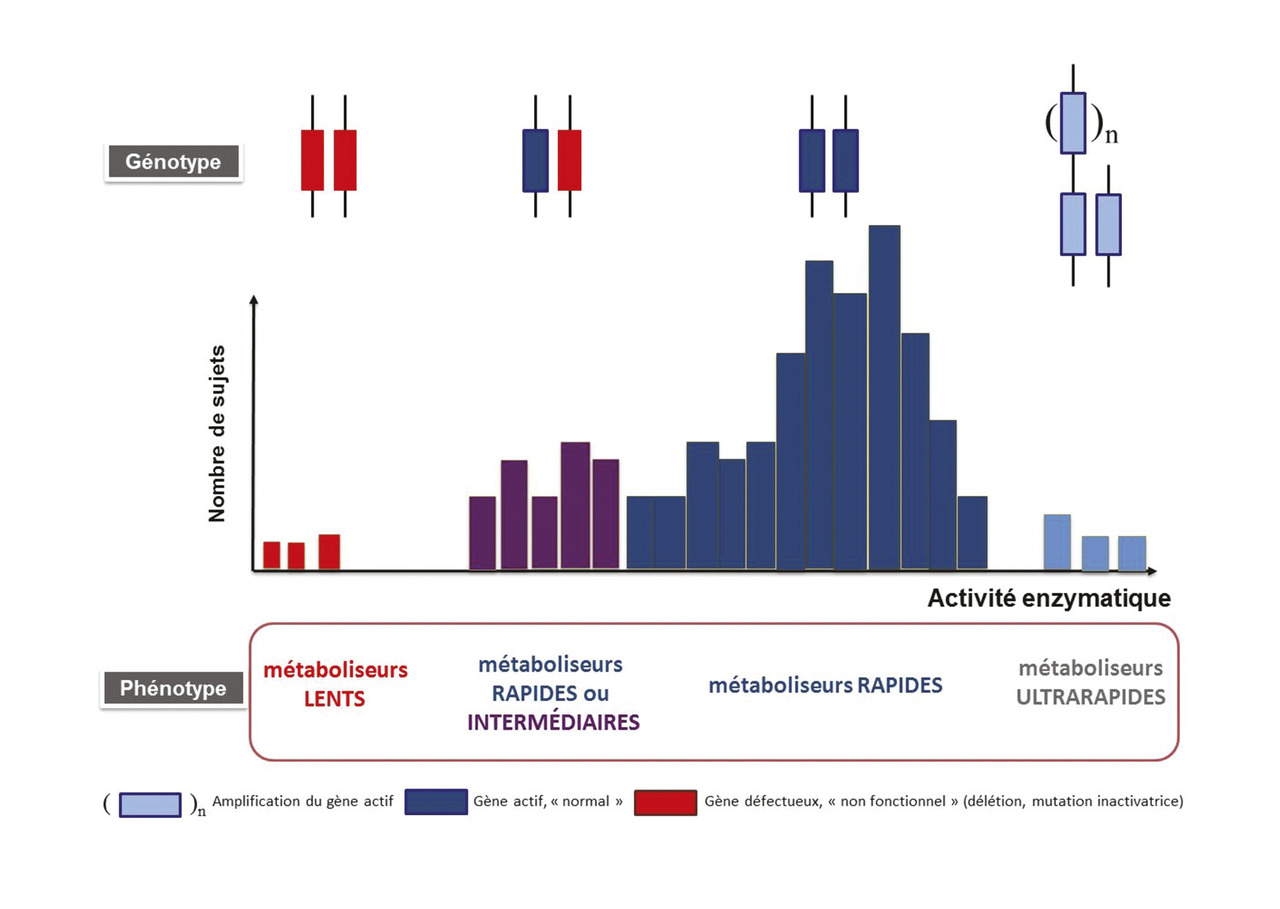
Différents sous-groupes de métaboliseurs
La plupart des gènes (métabolisme, transport, récepteurs) sont susceptibles d’avoir un rôle déterminant dans la réponse aux médicaments. En pratique, les applications de la pharmacogénétique concernent majoritairement les enzymes du métabolisme des médicaments. Les gènes codant ces enzymes peuvent présenter des variations de séquences, telles que des mutations ponctuelles ou single nucleotide polymorphism (SNP), des délétions partielles ou totales, ou encore des duplications ou amplifications de gènes. L’existence dans la population générale d’allèles différents pour un même gène, et, par conséquent, de différents génotypes, définit un polymorphisme génétique. Les polymorphismes génétiques peuvent être responsables de variations d’expression et/ou d’activité de ces enzymes, entraînant un déficit ou une absence totale d’activité ou encore une augmentation de l’activité. Les conséquences cliniques de ces variations génétiques dépendent d’un certain nombre de facteurs : importance de la voie métabolique polymorphe dans la clairance globale du médicament, administration du médicament sous une forme active ou d’un précurseur inactif, métabolites pharmacologiquement actifs ou non, métabolites toxiques ou non, indice thérapeutique du médicament.
Les polymorphismes génétiques des enzymes du métabolisme des médicaments s’expriment dans la population générale sous la forme de phénotypes définissant différents sous-groupes (v. figure ) :
– les métaboliseurs rapides (activité enzymatique normale) porteurs d’un génotype homozygote sauvage ;
– les métaboliseurs lents (déficit profond ou complet d’activité enzymatique) porteurs d’un génotype homozygote muté ou hétérozygote composite entraînant une perte de fonction du gène ;
– les métaboliseurs intermédiaires (activité enzymatique réduite) porteurs d’un génotype hétérozygote ;
– dans certains cas, on peut identifier des métaboliseurs ultrarapides (activité augmentée) porteurs le plus souvent d’une duplication ou amplification d’un gène fonctionnel. La fréquence de ces différents phénotypes est variable dans la population en fonction de l’enzyme polymorphe et, pour une même enzyme, elle varie en fonction de l’origine ethnique ou géographique des populations étudiées en raison de différences dans la nature et la fréquence des polymorphismes rencontrés.2
Les polymorphismes génétiques des enzymes du métabolisme des médicaments s’expriment dans la population générale sous la forme de phénotypes définissant différents sous-groupes (
– les métaboliseurs rapides (activité enzymatique normale) porteurs d’un génotype homozygote sauvage ;
– les métaboliseurs lents (déficit profond ou complet d’activité enzymatique) porteurs d’un génotype homozygote muté ou hétérozygote composite entraînant une perte de fonction du gène ;
– les métaboliseurs intermédiaires (activité enzymatique réduite) porteurs d’un génotype hétérozygote ;
– dans certains cas, on peut identifier des métaboliseurs ultrarapides (activité augmentée) porteurs le plus souvent d’une duplication ou amplification d’un gène fonctionnel. La fréquence de ces différents phénotypes est variable dans la population en fonction de l’enzyme polymorphe et, pour une même enzyme, elle varie en fonction de l’origine ethnique ou géographique des populations étudiées en raison de différences dans la nature et la fréquence des polymorphismes rencontrés.2
Des tests sous-utilisés
La pharmacogénétique vise principalement les médicaments ayant un indice thérapeutique étroit, pour lesquels l’efficacité thérapeutique est difficile à évaluer rapidement, ou encore ceux pour lesquels le médecin dispose de plusieurs alternatives thérapeutiques avec des profils de tolérance variable (tableaux 1 et 2 ). Quelques exemples permettent d’illustrer l’impact thérapeutique de cet outil de prédiction de la réponse aux médicaments. Des accidents toxiques graves ayant des conséquences économiques lourdes (hospitalisations, traitements coûteux mis en œuvre) pouvant conduire au décès ont été rapportés en cas de traitement par les thiopurines (leucémies et maladies auto-immunes) et de déficit en thiopurine S-méthyltransférase (TPMT), pour les malades ayant un déficit en dihydropyrimidine déshydrogénase (DPYD) et recevant du 5-fluoro-uracile (très largement utilisé pour le traitement des tumeurs solides), ou pour ceux atteints de la maladie de Gilbert (déficit en UDP-glucuronosyltransférase [UGT1A1]) et traités par l’irinotécan (cancers digestifs). Les « pro-médicaments », dont l’efficacité repose sur la transformation en composé actif, elle-même sous la dépendance d’une enzyme ayant un polymorphisme dans la population (cas du CYP2D6 et du tamoxifène prescrit dans les cancers du sein), sont également de bons exemples pour démontrer l’intérêt de la pharmacogénétique.3
La prescription des tests de pharmacogénétique dépend pour une large part de recommandations des sociétés savantes des biologistes. En France, le Réseau national de pharmacogénétique (RNPGx) et le Groupe de pharmacologie clinique oncologique (GPCO)-Unicancer regroupent les professionnels réalisant ces analyses avec pour principaux objectifs l’harmonisation des pratiques et la diffusion de recommandations fondées sur un bon niveau de preuves.4 Ces recommandations sont en accord avec celles publiées ailleurs en Europe ou par le consortium international Clinical Pharmacogenetics Implementation Consortium (CPIC). L’actualité récente a vu se concrétiser les efforts d’intégration de la pharmacogénétique dans le soin courant. Ainsi, l’obligation légale de réaliser un test avant l’initiation d’un traitement concerne désormais plusieurs médicaments, dont l’abacavir (allèle HLA*B5704), l’éliglustat (gène CYP2D6) et, depuis décembre 2018, les fluoropyrimidines (5-FU, capécitabine) (gène DPYD), tandis qu’une modification du résumé des caractéristiques de l’Imurel par l’Agence nationale de sécurité du médicament (ANSM) en 2018 préconise le dépistage du déficit en TPMT avant l’initiation du traitement. Cependant, si aujourd’hui près de 25 000 patients par an bénéficient d’un examen de pharmacogénétique selon les activités déclarées à l’Agence de la biomédecine par les laboratoires réalisant ces tests (soit environ 6 % de l’activité nationale de génétique moléculaire), ces chiffres attestent que le nombre de patients dépistés reste très largement inférieur à celui de la population cible recevant les traitements concernés.
La prescription des tests de pharmacogénétique dépend pour une large part de recommandations des sociétés savantes des biologistes. En France, le Réseau national de pharmacogénétique (RNPGx) et le Groupe de pharmacologie clinique oncologique (GPCO)-Unicancer regroupent les professionnels réalisant ces analyses avec pour principaux objectifs l’harmonisation des pratiques et la diffusion de recommandations fondées sur un bon niveau de preuves.4 Ces recommandations sont en accord avec celles publiées ailleurs en Europe ou par le consortium international Clinical Pharmacogenetics Implementation Consortium (CPIC). L’actualité récente a vu se concrétiser les efforts d’intégration de la pharmacogénétique dans le soin courant. Ainsi, l’obligation légale de réaliser un test avant l’initiation d’un traitement concerne désormais plusieurs médicaments, dont l’abacavir (allèle HLA*B5704), l’éliglustat (gène CYP2D6) et, depuis décembre 2018, les fluoropyrimidines (5-FU, capécitabine) (gène DPYD), tandis qu’une modification du résumé des caractéristiques de l’Imurel par l’Agence nationale de sécurité du médicament (ANSM) en 2018 préconise le dépistage du déficit en TPMT avant l’initiation du traitement. Cependant, si aujourd’hui près de 25 000 patients par an bénéficient d’un examen de pharmacogénétique selon les activités déclarées à l’Agence de la biomédecine par les laboratoires réalisant ces tests (soit environ 6 % de l’activité nationale de génétique moléculaire), ces chiffres attestent que le nombre de patients dépistés reste très largement inférieur à celui de la population cible recevant les traitements concernés.
Quelles pistes de développement ?
Les freins au déploiement à large échelle des tests de pharmacogénétique sont d’origine multifactorielle. En dehors des aspects réglementaires incontournables (recueil du consentement signé, diffusion très contrainte des résultats de génétique), d’un défaut de connaissances de l’utilité des tests et de leur interprétation, et de l’accès parfois difficile à ces tests pour les professionnels de santé (médecin, biologiste et pharmacien hospitalier), les difficultés rencontrées concernent plutôt le financement des actes (non remboursés par la Sécurité sociale dans la plupart des cas) et l’adaptation aux nouvelles technologies qui permettraient de couvrir de manière exhaustive l’ensemble des variants potentiellement délétères.5 L’évolution majeure de la pharmacogénétique va reposer sur la possibilité de réaliser des recherches élargies avec des outils de séquençage à haut débit, grâce à la création de plateformes génomiques à visée diagnostique et de suivi thérapeutique (les deux premières, SeQOIA et AURAGEN, étant implantées en Île-de-France et Auvergne-Rhône-Alpes) dans le cadre du plan France Médecine génomique 2025. La mise en place d’un « passeport pharmacogénétique » accessible aux professionnels de santé assurant la prise en charge thérapeutique tout au long de la vie du patient pourrait bouleverser la prescription des médicaments et sécuriser leur utilisation.6, 7 Enfin, l’intelligence artificielle favorise l’émergence de nouveaux outils numériques, fondés sur des algorithmes décisionnels intégrant des données génétiques afin d’adapter les doses de médicaments et l’espacement des prises en fonction des informations fournies directement par le patient, et communiquant en temps réel avec le dossier informatisé du patient, pouvant ainsi, le cas échéant, alerter le médecin en charge du patient.
Éviter une perte de chance
La pharmacogénétique offre l’opportunité de développer une médecine de précision hautement prédictive avec pour conséquence la diminution de l’incidence et du coût de l’iatrogénie médicamenteuse. Le prescripteur peut dès lors se poser la question d’un point de vue éthique : comment justifier a posteriori un accident toxique alors qu’il existe des moyens de prédire sa survenue ? Des patients ne bénéficient pas actuellement de la recherche de leurs caractéristiques génétiques susceptibles d’être à l’origine d’une toxicité ou d’une inefficacité de leur traitement médicamenteux, alors qu’elle est réalisable, ce qui constitue une perte de chance pour le patient et met en jeu la responsabilité des médecins et des autorités de tutelle. Du décryptage de l’ADN de l’homme de Néandertal à l’intégration des données génétiques dans des objets connectés, l’optimisation thérapeutique fondée sur la pharmacogénétique continue d’emprunter des voies singulières pour parvenir à son but ultime de sécuriser l’usage des médicaments !
Références
1. Vernot B, Akey JM. Resurrecting surviving Neanderthal lineages from modern human genomes. Science 2014;343:1017-21.
2. Krol A, Beaune P, de Waziers I, Loriot MA. Métabolisme des xénobiotiques. EMC Hépatologie (Elsevier Masson SAS, Paris) 2015 ;1976(15).
3. Daly AK. Pharmacogenetics: a general review on progress to date. British Med Bull 2017:124:65-79.
4. Picard N, Boyer JC, Etienne-Grimaldi MC, Barin-Le Guellec C, Thomas F, Loriot MA. Pharmacogenetics-based personalized therapy: Levels of evidence and recommendations from the French Network of Pharmacogenetics (RNPGx). Thérapie 2017;72:175-83.
5. Ingelman-Sundberg M, Mkrtchian S, Zhou Y, Lauschke VM. Integrating rare genetic variants into pharmacogenetics drug response predictions. Human Genomics 2018;12:26.
6. Caraballo PJ, Bielinski SJ, St Sauver JL, Weinshilboum RM. Electronic medical record-integrated pharmacogenomics and related clinical decision support concepts. Clin Pharmacol Ther 2017;102:254-64.
7. Van der Wouden CH, van Rhenen MH, et al. Development of the PGx-passport: a panel of actionable germline genetic variants for pre-emptive pharmacogenetic testing. Clin Pharmacol Ther 2019;106:866-73.
2. Krol A, Beaune P, de Waziers I, Loriot MA. Métabolisme des xénobiotiques. EMC Hépatologie (Elsevier Masson SAS, Paris) 2015 ;1976(15).
3. Daly AK. Pharmacogenetics: a general review on progress to date. British Med Bull 2017:124:65-79.
4. Picard N, Boyer JC, Etienne-Grimaldi MC, Barin-Le Guellec C, Thomas F, Loriot MA. Pharmacogenetics-based personalized therapy: Levels of evidence and recommendations from the French Network of Pharmacogenetics (RNPGx). Thérapie 2017;72:175-83.
5. Ingelman-Sundberg M, Mkrtchian S, Zhou Y, Lauschke VM. Integrating rare genetic variants into pharmacogenetics drug response predictions. Human Genomics 2018;12:26.
6. Caraballo PJ, Bielinski SJ, St Sauver JL, Weinshilboum RM. Electronic medical record-integrated pharmacogenomics and related clinical decision support concepts. Clin Pharmacol Ther 2017;102:254-64.
7. Van der Wouden CH, van Rhenen MH, et al. Development of the PGx-passport: a panel of actionable germline genetic variants for pre-emptive pharmacogenetic testing. Clin Pharmacol Ther 2019;106:866-73.