La parution dans Nature et Science en 2001 de deux brouillons de la séquence du génome humain était l’aboutissement d’un travail gigantesque qui avait mobilisé pendant de nombreuses années des efforts intellectuels et financiers, sans équivalent jusque-là dans le monde de la biologie et qu’on a comparé pour cette discipline à la conquête de la lune en 1969.
Les 15 et 16 février 2001 paraissaient dans le numéro 6 822 de Nature1 et le numéro 5 507 de Science2 deux brouillons de la séquence du génome humain publiés le premier par un consortium international regroupant 20 laboratoires de 6 pays et plusieurs centaines d’auteurs sous la houlette d’Éric Lander et le second sous la direction du biologiste entrepreneur Craig Venter, créateur de la Biotech Celera Genomics (fig. 1 ).
Les premiers pas
Pour comprendre l’importance qu’a revêtu cet exploit qui en réalité n’était qu’une étape, il convient de retracer ne serait-ce que brièvement l’histoire de l’ADN. C’est au xixe siècle, en 1869, que Johann Friedrich Miescher, biologiste suisse, identifia dans les noyaux des cellules la nucléine appelée acide déosyribonucléique ou ADN. Il faudra toutefois attendre les travaux de Thomas Hunt Morgan pour affirmer le rôle des chromosomes dans l’hérédité, ce qui lui valut en 1933 d’être honoré par le prix Nobel de médecine, et ceux de Frederick Griffith en 1928 et Oswald Avery en 1944 pour définitivement statuer sur l’importance primordiale de l’ADN dans l’hérédité. L’histoire s’accélère encore avec la proposition par James Watson et Francis Crick en 1953 du modèle structural en double hélice de l’ADN fondé sur l’interprétation des travaux de cristallographie de Rosalind Franklin, mais aussi d’Erwin Chargaff montrant la parité adénine/thymine d’une part et guanine/cytosine d’autre part dans toutes les molécules d’ADN.
Au pied du mur
C’est au tournant des années 1968-1969 que Frederick Sanger a commencé sérieusement à s’intéresser au séquençage de l’ADN (fig. 2 ). Préalablement à son implication dans ce domaine Sanger avait été un pionnier du séquençage des macromolécules. En premier, ce fut le développement d’une méthode originale d’analyse de la structure primaire des protéines qui, avec la détermination de celle de l’insuline, lui valut en 1958 son premier prix Nobel de chimie. Après ce premier coup d’éclat, il orienta ses recherches vers l’analyse de la structure primaire des ARN, domaine où il fut tout aussi prolifique en développant une méthode de séquençage originale et fiable qui fut à la base de l’analyse de centaines de molécules d’ARN de transfert, mais aussi de génomes de virus à ARN, en particulier de phages. Ce n’est que par un malheureux concours de circonstances qu’il ne fut pas le premier à déterminer la séquence d’un ARN de transfert (ARNt). Cet honneur revint à William Robert Holley qui, le premier après un travail titanesque, décrivit la structure primaire de l’ARNt de l’alanine et reçut pour cela et son apport au mécanisme de synthèse des protéines le prix Nobel de médecine en 1958 partagé avec Marshall W. Nirenberg et Har G. Khorana. Mais, détail piquant, la méthode développée pour le séquençage de l’ARNt de l’alanine proposée par Holley ne fut jamais appliquée à d’autres molécules tant elle était lourde (rappelons brièvement qu’elle nécessita 1 g d’ARNt purifié à partir de 140 kg de levure) et globalement peu efficiente à la différence de celle de Sanger. Après les protéines et les ARN, Sanger s’est donc intéressé à l’ADN. La question qui surgit immédiatement est de comprendre pourquoi il avait privilégié de décrire dans l’ordre protéine, ARN puis ADN dans la mesure où toutes les informations nécessaires à la synthèse des protéines sont contenues dans l’ADN et que, sachant déterminer la séquence de l’ADN, on peut théoriquement en déduire celles des ARN et les protéines. Sanger s’en expliqua dans le discours de réception de son deuxième prix Nobel :
« In spite of the important role played by DNA sequences in living matter, it is only relatively recently that general methods for their determination have been developed. This is mainly because of the very large size of DNA molecules, the smallest being those of the simple bacteriophages such as Phi X 174 (which contains about 5,000 nucleotides). It was therefore difficult to develop methods with such complicated systems. There are however some relatively small RNA molecules - notably the transfer RNAs of about 75 nucleotides, and these were used for the early studies on nucleic acid sequences. »
Et, de fait, lorsque en 1969 le petit groupe animé par Sanger commença à s’investir dans le développement d’une méthode d’analyse de l’ADN, on ne disposait d’aucun fragment d’ADN de petite taille, ni d’enzyme pour digérer l’ADN comme cela avait été le cas pour la mise au point des méthodes de séquençage des autres macromolécules. Les enzymes de restrictions (ER) n’existaient pas encore. Leur existence fut prédite par Werner Arber3 en 1962 et la première isolée, purifiée et caractérisée, Hinc II, ne le fut qu’au début des années 1970.4, 5
Il est en effet primordial du point de vue épistémologique de bien comprendre le caractère quasi insoluble des défis devant lequel nous nous trouvions : pas de molécule d’ADN susceptible de servir de modèle, pas d’enzyme pour la découper en fragments de plus en plus petits dont la séquence individuelle deviendrait évidente et réassemblage de ces sous-éléments à l’image de ce qui avait été fait pour les protéines avec l’insuline ou les ARN de transfert et l’ARN ribosomique 5S.
Les tous premiers résultats significatifs dans le domaine sont dus à Ray Wu qui, en 1968, détermina la séquence des 12 nucléotides de l’extrémité cohésive du phage lambda.6 Quoique modeste, ce résultat, on le verra plus tard, montrant la possibilité d’extension à partir de l’extrémité 3’ d’une chaîne d’ADN, fût en réalité très important, il conditionnera tout le développement futur du séquençage de l’ADN.
Dans le laboratoire de Sanger, les premiers essais se réduisirent à traiter l’ADN monocaténaire du phage par des solutions acides et à détruire ainsi spécifiquement toutes les bases puriques pour ne laisser subsister que les déoxyoligonucléotides constitués uniquement de bases pyrimidiques. Dans les expériences réalisées par Vic Ling, post-doctorant du groupe à partir du génome du phage fd des oligonucléotides de taille très variable allant jusqu’à une vingtaine de cytosines et de thymines furent obtenus. Après purification de ces dernières, leur enchaînement précis fut déterminé par des hydrolyses ménagées avec des phosphodiestérases digérant séquentiellement l’ADN à partir de son extrémité 3’ ou 5’. Résultat bien modeste mais montrant bien l’immensité de la tâche qui restait à accomplir.7
Après ces premiers essais, ce fut le génome du phage monocaténaire de Phi X174 qui fut choisi sans raison particulière par rapport à d’autres possibles, si ce n’est que ce phage avait été identifié par John Sedat au cours de sa thèse faite chez Robert Sinsheimer en Californie et que John faisait partie de notre petite équipe. Le premier objectif fut de trouver un moyen d’obtenir, à partir du génome complet de Phi X 174, des fragments purs de petite taille. Deux stratégies furent suivies :
– l’une d’elle animée par Hugh Robertson, post-doctorant américain, consista à utiliser des ribosomes bactériens pour se fixer sur le génome monocaténaire de phiX174 à l’image d’un complexe ribosomique se fixant spécifiquement sur un ARN messager pour initier la traduction du message. L’action de la Dnase I après fixation spécifique du ribosome sur l’ADN de phage permit de détruire tout l’ADN non protégé ;
– l’autre, plus générique dans son principe, fut d’utiliser les propriétés d’une Dnase, l’Endo IV, qui a une double préférence. Elle hydrolyse les liaisons C-N de préférence aux autres dans les régions monocaténaires. Or l’ADN de phiX174 en solution saline concentrée adopte majoritairement une structure double brin occasionnée par des repliements sur elle-même, laissant des zones monocaténaires, dès lors accessible à l’Endo IV. C’est ainsi qu’en solution hypertonique plusieurs fragments de 100 nucléotides environ ont pu être préparés. L’hydrolyse avec l’Endo IV en solution hypotonique de ces fragments purifiés nous a permis d’obtenir une vingtaine d’oligonucléotides dont la séquence a été déterminée en appliquant autant que faire se peut les méthodes parfaitement maitrisées de séquençage de l’ARN. Pour mener à bien ces opérations de l’ADN de phiX174 était préparé à partir de culture d’Escherichia coli infectée et cultivée en présence de 100 millicuries d’orthophospate 32P, quantité de radioactivité qu’il ne serait plus possible d’utiliser aujourd’hui dans un laboratoire de biologie. C’est ainsi qu’un fragment d’ADN de 48 nucléotides fut entièrement séquencé pour la première fois.8 Par la suite plusieurs fragments de plus grande taille ont été produits et séquencés.
Considérant toutefois que le verrou principal était désormais l’obtention de fragments d’ADN dans la mesure où nous avions pu développer une méthode, certes très laborieuse mais efficace d’analyse de ceux-ci, Sanger, s’inspirant des travaux de R. Wu,6 rechercha et développa une autre approche de préparation. Ayant remarqué que les positions 26 à 28 de la séquence de la protéine d’enveloppe du virus fd était Trp-Met-Val, on pouvait en déduire que ce tri-peptide correspondait à la séquence TGAATGGG. L’oligonucléotide complémentaire fut donc synthétisé, hybridé à l’ADN de phage et servit d’amorce pour la synthèse in vitro de molécules d’ADN en présence d’ADN polymérase et de déoxynucléoside triphosphates (dNTP) marqués au 32P. L’analyse par les méthodes que nous avions préalablement développées des produits ainsi obtenus montrèrent que la synthèse correspondait à un site d’initiation unique démontrant que l’approche était propice à la préparation de fragments tant souhaités d’ADN.9
Poursuivant son travail, Sanger eut idée d’ajouter au milieu de synthèse en plus des 4 (dNTP) un ribonucléoside triphosphate (rNTP). Lequel s’incorporait dans la chaîne d’ADN en cours d’élongation en lieu et place de son homologue dNTP en proportion de leur concentration respective dans le milieu réactionnel. La réalisation en parallèle de quatre réactions faites chacune en présence d’un rNTP différent fournit un jeu de molécules d’ADN qui, après traitement par une ribonucléase hydrolysant les produits de synthèse au niveau du ribonucléotide (rN) incorporé, fournit quatre jeux de molécules plus ou moins longues d’ADN débutant toutes à la position 5’ de l’amorce. La migration électrophorétique de ces molécules en gel d’acrylamide permet de lire directement sur l’autoradiogramme la séquence en notant la travées A, G, C o u T où se trouve la bande la plus mobile correspondant à la plus petite molécule, puis de noter le nom de la travée dans laquelle se trouve la molécule de mobilité immédiatement inférieure et correspondant à la molécule n+1. En nommant échelon après échelon la position des bandes successives on écrit directement la séquence de la copie complémentaire néosynthétisée de la séquence à analyser.
Sanger imagina ensuite la méthode « plus and minus », variante de la précédente, dans laquelle après une synthèse brève fournissant à partir de l’amorce un jeu de molécules de longueur aléatoire, la synthèse était reprise dans quatre tubes dans lesquels était ajouté un seul dNTP. Ainsi, dans le tube +A où n’était ajouté que de l’adénosine triphosphate, la synthèse ne pouvait reprendre que si elle s’était arrêtée juste avant l’incorporation de ce dernier. Si ce n’était pas le cas, la polymérase ne pouvant poursuivre la synthèse mettait en jeu sa capacité de dégradation de l’ADN de façon séquentielle à partir de l’extrémité 3’ de la molécule. Cette action se poursuivait jusqu’à la présence d’une thymine sur la chaîne à copier. Dès lors, un équilibre se faisait entre la capacité de synthèse de la polymérase et sa capacité d’hydrolyse, la résultante étant une molécule d’ADN néosynthétisée se terminant par A. Dans les trois autres tubes s’accumulaient, pour les mêmes raisons, des molécules se terminant par G, C ou T. La partie « minus » de la méthode n’était en quelque sorte que le symétrique de la moitié « plus ». En effet, dans cette partie, la synthèse était reprise en parallèle dans quatre tubes, mais cette fois on jouxtait trois dNTP. Pour les mêmes raisons que précédemment la synthèse reprenait mais s’arrêtait juste avant l’incorporation du dNTP manquant. La migration électrophorétique en parallèle dans un gel d’acrylamide des huit groupes de molécules permettait de lire à partir de la mobilité des différentes molécules la séquence recherchée. Les lectures indépendantes des parties plus et moins devaient assurer la justesse du résultat. Seules les régions homopolymères de plus de deux résidus étaient incomplètement analysées, apparaissant sur l’autoradiogramme sous la forme d’un espace vide plus ou moins grand en fonction de la longueur de l’homopolymère.
Bien que complexe dans sa description, cette méthode, déjà beaucoup plus puissante que les précédentes, permit la détermination de la séquence complète du génome de phiX174 de quelque 5 375 nucléotides dont l’interprétation de la position des phases ouvertes codant les huit gènes du phage montra que, de façon surprenante car totalement inédite, plusieurs gènes étaient en partie « chevauchants », c’est-à-dire qu’ils étaient codés pour une partie par la même séquence de nucléotides mais lus par la machinerie cellulaire dans deux phases différentes.10 Pour la détermination de cette séquence, le problème clé de l’amorçage de la néosynthèse d’ADN fut résolu en l’absence d’oligonucléotides de séquences définies impossibles à obtenir à l’époque aisément par l’hybridation de fragments d’ADN chevauchant obtenus avec les quelques enzymes de restriction (ER) qui commencèrent à être disponibles à partir de 1974.
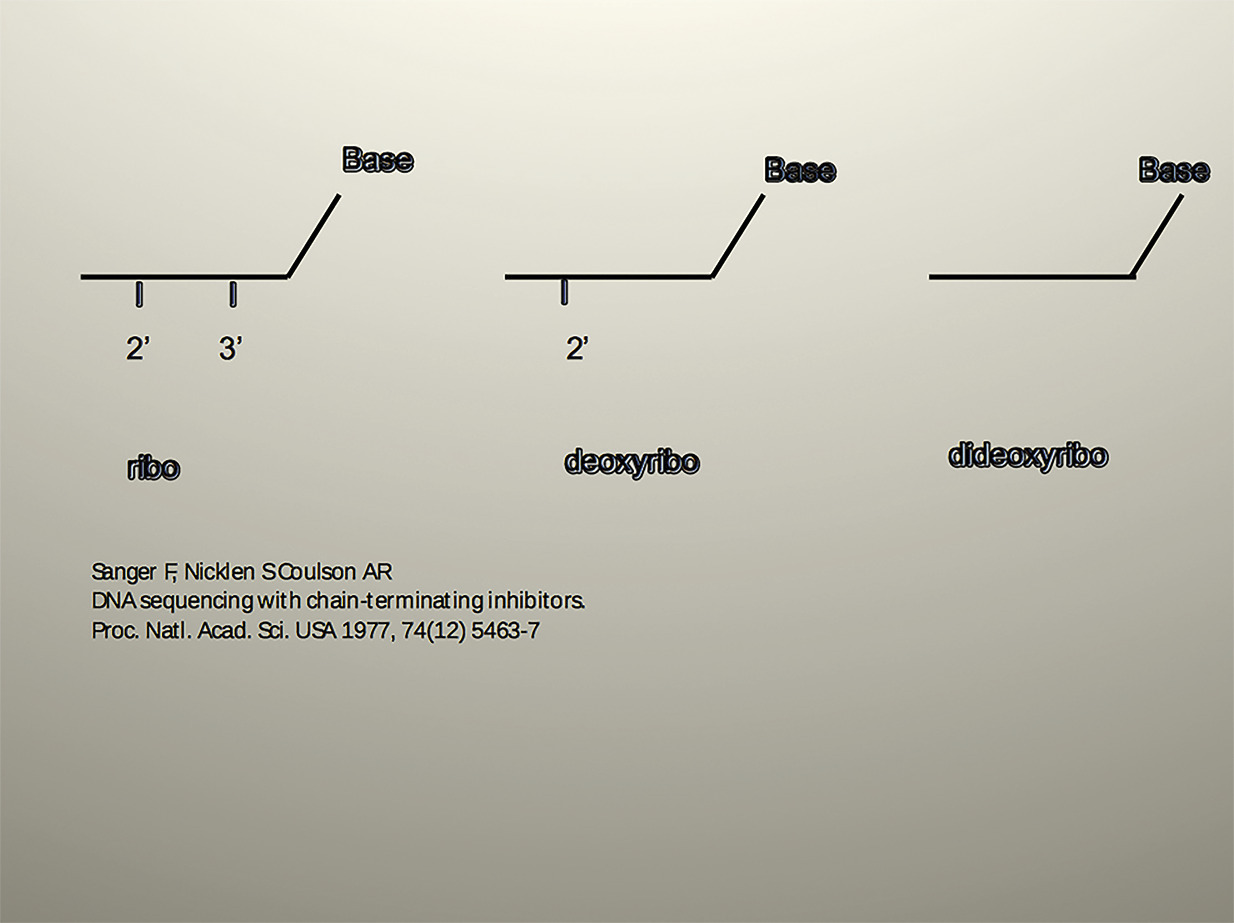
La quête d’une méthode encore plus performante et qui, en particulier, résoudrait le problème posé par les homopolymères dont la taille ne pouvait être déterminée par la méthode « plus-moins » déboucha sur la méthode des terminaisons de chaînes ou ddN (ou ddNTP), qui n’est qu’une variante des méthodes précédentes, la ribosubstitution et la méthode « plus-moins », dans la mesure où, dans ce cas, l’arrêt de la synthèse est obtenu par l’incorporation d’un dérivé ddNTP dépourvu d’hydroxyle en 2’ et 3’(fig. 3 ). Toutefois, le changement est doublement important dans la mesure où, dès le début, les ddNTP sont ajoutés en proportion adéquate par rapport aux dNTP, ce qui évite la phase préalable d’élongation et surtout donne des arrêts plus francs se traduisant par une lecture plus facile et fiable des autoradiogrammes.11
Parallèlement au travail réalisé à Cambridge (UK), Walter Gilbert, biologiste à la Harvard Medical School, s’intéressait à la régulation de l’expression de l’opéron lactose d’Escherichia. coli. À la différence de Sanger, son souhait n’était pas de développer une méthode générique de séquençage de l’ADN mais de décrypter les mécanismes intimes de la régulation de l’opéron lactose d’E. coli. Pour cela il développa une stratégie similaire à celle de Hugh Roberstson, consistant à protéger l’opérateur par la fixation spécifique de son répresseur et à digérer tout le reste du DNA génomique par de la Dnase pancréatique. La résultante de cette opération fut un fragment d’ADN double brin dont l’analyse de la séquence par différentes approches dérivées pour partie des méthodes de séquençage de l’ARN permirent la déduction de sa séquence de 24 nucléotides.12
Mettant à profit la commercialisation récente de quelques enzymes de restriction, W. Gilbert et Allan M. Maxam préparèrent un fragment de restriction de plus grande taille avec l’enzyme Alu I qui hydrolyse l’ADN double brin au milieu du tétranucléotide AGCT. Le principe de la méthode de séquençage développée par et W. Gilbert repose sur une série de réactions chimiques. La première consiste à traiter l’ADN par un réactif comme l’hydrazine dans des conditions limites qui vont modifier de façon spécifique un certain nombre de pyrimidines et permettre leur élimination de la chaîne d’ADN. La conséquence de cette élimination est une fragilisation des liaisons phosphodiesters en 3’ qui peuvent être alors rompues. L’utilisation en parallèle de réactifs chimiques affectant d’autres bases comme le diméthyl sulfate qui méthyle en 7’ les bases puriques permet d’obtenir des molécules d’ADN qui se terminent avec le résidu précédant la base attaquée par le réactif chimique.
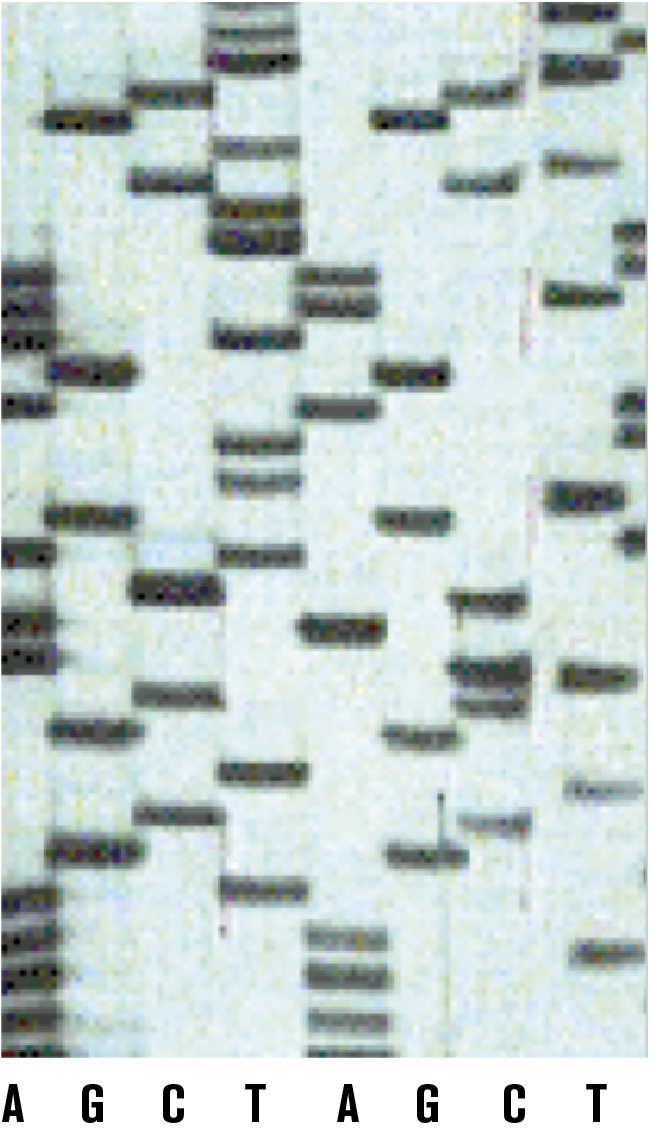
Le fragment de restriction à séquencer est d‘abord déphosphorylé puis marqué avec du 32P en position 5’. Après marquage, une électrophorèse en gel d’acrylamide permet de séparer les deux chaînes. Chaque chaîne est alors traitée indépendamment par les différents réactifs et les produits résultant des différentes réactions séparées par électrophorèse en gel d’acrylamide. Comme dans le cas de la méthode aux DDN, l’autoradiogramme du gel révèle la position des différents fragments obtenus qui tous débutent à partir de l’extrémité 5’ marquée au 32P pour se terminer sur la base qui précède celle endommagée chimiquement.
Ici encore l’interprétation de la mobilité des différentes bandes donne la séquence du fragment traité (fig. 4 ). La lecture indépendante des résultats obtenus avec l’autre chaîne complémentaire confirme de façon non ambiguë les résultats.13
Ces deux méthodes publiées en 1977 à quelques mois d’intervalle ont bouleversé nos capacités d’analyse d’abord en médecine, puis dans beaucoup d’autres domaines, compte tenu de la présence ubiquitaire de l’ADN.
« In spite of the important role played by DNA sequences in living matter, it is only relatively recently that general methods for their determination have been developed. This is mainly because of the very large size of DNA molecules, the smallest being those of the simple bacteriophages such as Phi X 174 (which contains about 5,000 nucleotides). It was therefore difficult to develop methods with such complicated systems. There are however some relatively small RNA molecules - notably the transfer RNAs of about 75 nucleotides, and these were used for the early studies on nucleic acid sequences. »
Et, de fait, lorsque en 1969 le petit groupe animé par Sanger commença à s’investir dans le développement d’une méthode d’analyse de l’ADN, on ne disposait d’aucun fragment d’ADN de petite taille, ni d’enzyme pour digérer l’ADN comme cela avait été le cas pour la mise au point des méthodes de séquençage des autres macromolécules. Les enzymes de restrictions (ER) n’existaient pas encore. Leur existence fut prédite par Werner Arber3 en 1962 et la première isolée, purifiée et caractérisée, Hinc II, ne le fut qu’au début des années 1970.4, 5
Il est en effet primordial du point de vue épistémologique de bien comprendre le caractère quasi insoluble des défis devant lequel nous nous trouvions : pas de molécule d’ADN susceptible de servir de modèle, pas d’enzyme pour la découper en fragments de plus en plus petits dont la séquence individuelle deviendrait évidente et réassemblage de ces sous-éléments à l’image de ce qui avait été fait pour les protéines avec l’insuline ou les ARN de transfert et l’ARN ribosomique 5S.
Les tous premiers résultats significatifs dans le domaine sont dus à Ray Wu qui, en 1968, détermina la séquence des 12 nucléotides de l’extrémité cohésive du phage lambda.6 Quoique modeste, ce résultat, on le verra plus tard, montrant la possibilité d’extension à partir de l’extrémité 3’ d’une chaîne d’ADN, fût en réalité très important, il conditionnera tout le développement futur du séquençage de l’ADN.
Dans le laboratoire de Sanger, les premiers essais se réduisirent à traiter l’ADN monocaténaire du phage par des solutions acides et à détruire ainsi spécifiquement toutes les bases puriques pour ne laisser subsister que les déoxyoligonucléotides constitués uniquement de bases pyrimidiques. Dans les expériences réalisées par Vic Ling, post-doctorant du groupe à partir du génome du phage fd des oligonucléotides de taille très variable allant jusqu’à une vingtaine de cytosines et de thymines furent obtenus. Après purification de ces dernières, leur enchaînement précis fut déterminé par des hydrolyses ménagées avec des phosphodiestérases digérant séquentiellement l’ADN à partir de son extrémité 3’ ou 5’. Résultat bien modeste mais montrant bien l’immensité de la tâche qui restait à accomplir.7
Après ces premiers essais, ce fut le génome du phage monocaténaire de Phi X174 qui fut choisi sans raison particulière par rapport à d’autres possibles, si ce n’est que ce phage avait été identifié par John Sedat au cours de sa thèse faite chez Robert Sinsheimer en Californie et que John faisait partie de notre petite équipe. Le premier objectif fut de trouver un moyen d’obtenir, à partir du génome complet de Phi X 174, des fragments purs de petite taille. Deux stratégies furent suivies :
– l’une d’elle animée par Hugh Robertson, post-doctorant américain, consista à utiliser des ribosomes bactériens pour se fixer sur le génome monocaténaire de phiX174 à l’image d’un complexe ribosomique se fixant spécifiquement sur un ARN messager pour initier la traduction du message. L’action de la Dnase I après fixation spécifique du ribosome sur l’ADN de phage permit de détruire tout l’ADN non protégé ;
– l’autre, plus générique dans son principe, fut d’utiliser les propriétés d’une Dnase, l’Endo IV, qui a une double préférence. Elle hydrolyse les liaisons C-N de préférence aux autres dans les régions monocaténaires. Or l’ADN de phiX174 en solution saline concentrée adopte majoritairement une structure double brin occasionnée par des repliements sur elle-même, laissant des zones monocaténaires, dès lors accessible à l’Endo IV. C’est ainsi qu’en solution hypertonique plusieurs fragments de 100 nucléotides environ ont pu être préparés. L’hydrolyse avec l’Endo IV en solution hypotonique de ces fragments purifiés nous a permis d’obtenir une vingtaine d’oligonucléotides dont la séquence a été déterminée en appliquant autant que faire se peut les méthodes parfaitement maitrisées de séquençage de l’ARN. Pour mener à bien ces opérations de l’ADN de phiX174 était préparé à partir de culture d’Escherichia coli infectée et cultivée en présence de 100 millicuries d’orthophospate 32P, quantité de radioactivité qu’il ne serait plus possible d’utiliser aujourd’hui dans un laboratoire de biologie. C’est ainsi qu’un fragment d’ADN de 48 nucléotides fut entièrement séquencé pour la première fois.8 Par la suite plusieurs fragments de plus grande taille ont été produits et séquencés.
Considérant toutefois que le verrou principal était désormais l’obtention de fragments d’ADN dans la mesure où nous avions pu développer une méthode, certes très laborieuse mais efficace d’analyse de ceux-ci, Sanger, s’inspirant des travaux de R. Wu,6 rechercha et développa une autre approche de préparation. Ayant remarqué que les positions 26 à 28 de la séquence de la protéine d’enveloppe du virus fd était Trp-Met-Val, on pouvait en déduire que ce tri-peptide correspondait à la séquence TGAATGGG. L’oligonucléotide complémentaire fut donc synthétisé, hybridé à l’ADN de phage et servit d’amorce pour la synthèse in vitro de molécules d’ADN en présence d’ADN polymérase et de déoxynucléoside triphosphates (dNTP) marqués au 32P. L’analyse par les méthodes que nous avions préalablement développées des produits ainsi obtenus montrèrent que la synthèse correspondait à un site d’initiation unique démontrant que l’approche était propice à la préparation de fragments tant souhaités d’ADN.9
Poursuivant son travail, Sanger eut idée d’ajouter au milieu de synthèse en plus des 4 (dNTP) un ribonucléoside triphosphate (rNTP). Lequel s’incorporait dans la chaîne d’ADN en cours d’élongation en lieu et place de son homologue dNTP en proportion de leur concentration respective dans le milieu réactionnel. La réalisation en parallèle de quatre réactions faites chacune en présence d’un rNTP différent fournit un jeu de molécules d’ADN qui, après traitement par une ribonucléase hydrolysant les produits de synthèse au niveau du ribonucléotide (rN) incorporé, fournit quatre jeux de molécules plus ou moins longues d’ADN débutant toutes à la position 5’ de l’amorce. La migration électrophorétique de ces molécules en gel d’acrylamide permet de lire directement sur l’autoradiogramme la séquence en notant la travées A, G, C o u T où se trouve la bande la plus mobile correspondant à la plus petite molécule, puis de noter le nom de la travée dans laquelle se trouve la molécule de mobilité immédiatement inférieure et correspondant à la molécule n+1. En nommant échelon après échelon la position des bandes successives on écrit directement la séquence de la copie complémentaire néosynthétisée de la séquence à analyser.
Sanger imagina ensuite la méthode « plus and minus », variante de la précédente, dans laquelle après une synthèse brève fournissant à partir de l’amorce un jeu de molécules de longueur aléatoire, la synthèse était reprise dans quatre tubes dans lesquels était ajouté un seul dNTP. Ainsi, dans le tube +A où n’était ajouté que de l’adénosine triphosphate, la synthèse ne pouvait reprendre que si elle s’était arrêtée juste avant l’incorporation de ce dernier. Si ce n’était pas le cas, la polymérase ne pouvant poursuivre la synthèse mettait en jeu sa capacité de dégradation de l’ADN de façon séquentielle à partir de l’extrémité 3’ de la molécule. Cette action se poursuivait jusqu’à la présence d’une thymine sur la chaîne à copier. Dès lors, un équilibre se faisait entre la capacité de synthèse de la polymérase et sa capacité d’hydrolyse, la résultante étant une molécule d’ADN néosynthétisée se terminant par A. Dans les trois autres tubes s’accumulaient, pour les mêmes raisons, des molécules se terminant par G, C ou T. La partie « minus » de la méthode n’était en quelque sorte que le symétrique de la moitié « plus ». En effet, dans cette partie, la synthèse était reprise en parallèle dans quatre tubes, mais cette fois on jouxtait trois dNTP. Pour les mêmes raisons que précédemment la synthèse reprenait mais s’arrêtait juste avant l’incorporation du dNTP manquant. La migration électrophorétique en parallèle dans un gel d’acrylamide des huit groupes de molécules permettait de lire à partir de la mobilité des différentes molécules la séquence recherchée. Les lectures indépendantes des parties plus et moins devaient assurer la justesse du résultat. Seules les régions homopolymères de plus de deux résidus étaient incomplètement analysées, apparaissant sur l’autoradiogramme sous la forme d’un espace vide plus ou moins grand en fonction de la longueur de l’homopolymère.
Bien que complexe dans sa description, cette méthode, déjà beaucoup plus puissante que les précédentes, permit la détermination de la séquence complète du génome de phiX174 de quelque 5 375 nucléotides dont l’interprétation de la position des phases ouvertes codant les huit gènes du phage montra que, de façon surprenante car totalement inédite, plusieurs gènes étaient en partie « chevauchants », c’est-à-dire qu’ils étaient codés pour une partie par la même séquence de nucléotides mais lus par la machinerie cellulaire dans deux phases différentes.10 Pour la détermination de cette séquence, le problème clé de l’amorçage de la néosynthèse d’ADN fut résolu en l’absence d’oligonucléotides de séquences définies impossibles à obtenir à l’époque aisément par l’hybridation de fragments d’ADN chevauchant obtenus avec les quelques enzymes de restriction (ER) qui commencèrent à être disponibles à partir de 1974.
La quête d’une méthode encore plus performante et qui, en particulier, résoudrait le problème posé par les homopolymères dont la taille ne pouvait être déterminée par la méthode « plus-moins » déboucha sur la méthode des terminaisons de chaînes ou ddN (ou ddNTP), qui n’est qu’une variante des méthodes précédentes, la ribosubstitution et la méthode « plus-moins », dans la mesure où, dans ce cas, l’arrêt de la synthèse est obtenu par l’incorporation d’un dérivé ddNTP dépourvu d’hydroxyle en 2’ et 3’(
Parallèlement au travail réalisé à Cambridge (UK), Walter Gilbert, biologiste à la Harvard Medical School, s’intéressait à la régulation de l’expression de l’opéron lactose d’Escherichia. coli. À la différence de Sanger, son souhait n’était pas de développer une méthode générique de séquençage de l’ADN mais de décrypter les mécanismes intimes de la régulation de l’opéron lactose d’E. coli. Pour cela il développa une stratégie similaire à celle de Hugh Roberstson, consistant à protéger l’opérateur par la fixation spécifique de son répresseur et à digérer tout le reste du DNA génomique par de la Dnase pancréatique. La résultante de cette opération fut un fragment d’ADN double brin dont l’analyse de la séquence par différentes approches dérivées pour partie des méthodes de séquençage de l’ARN permirent la déduction de sa séquence de 24 nucléotides.12
Mettant à profit la commercialisation récente de quelques enzymes de restriction, W. Gilbert et Allan M. Maxam préparèrent un fragment de restriction de plus grande taille avec l’enzyme Alu I qui hydrolyse l’ADN double brin au milieu du tétranucléotide AGCT. Le principe de la méthode de séquençage développée par et W. Gilbert repose sur une série de réactions chimiques. La première consiste à traiter l’ADN par un réactif comme l’hydrazine dans des conditions limites qui vont modifier de façon spécifique un certain nombre de pyrimidines et permettre leur élimination de la chaîne d’ADN. La conséquence de cette élimination est une fragilisation des liaisons phosphodiesters en 3’ qui peuvent être alors rompues. L’utilisation en parallèle de réactifs chimiques affectant d’autres bases comme le diméthyl sulfate qui méthyle en 7’ les bases puriques permet d’obtenir des molécules d’ADN qui se terminent avec le résidu précédant la base attaquée par le réactif chimique.
Le fragment de restriction à séquencer est d‘abord déphosphorylé puis marqué avec du 32P en position 5’. Après marquage, une électrophorèse en gel d’acrylamide permet de séparer les deux chaînes. Chaque chaîne est alors traitée indépendamment par les différents réactifs et les produits résultant des différentes réactions séparées par électrophorèse en gel d’acrylamide. Comme dans le cas de la méthode aux DDN, l’autoradiogramme du gel révèle la position des différents fragments obtenus qui tous débutent à partir de l’extrémité 5’ marquée au 32P pour se terminer sur la base qui précède celle endommagée chimiquement.
Ici encore l’interprétation de la mobilité des différentes bandes donne la séquence du fragment traité (
Ces deux méthodes publiées en 1977 à quelques mois d’intervalle ont bouleversé nos capacités d’analyse d’abord en médecine, puis dans beaucoup d’autres domaines, compte tenu de la présence ubiquitaire de l’ADN.
L’embellie
Alors que Sanger avait cherché à développer une méthode générique et que Gilbert pensait surtout sinon exclusivement à l’opéron lactose, c’est la méthode des dégradations chimiques qui fut adoptée par la communauté scientifique et pas la méthode aux ddN. La raison en est très simple. La méthode de Gilbert était plus simple d’utilisation, donnait des lectures d’autoradiogrammes plus faciles et surtout elle était adaptée et efficace sur l’ADN double brin qui constitue l’essentiel de l’ADN sur terre, alors que celle de Sanger développée à partir du phage monocaténaire phiX174 fonctionnait difficilement avec l’ADN double brin. Cette préférence quasi absolue pour la méthode des dégradations chimiques s’est au cours des ans inversée au profit de la méthode aux ddN grâce à l’amélioration de la qualité des réactifs, mais surtout du développement par Joshua Messing d’un système de clonage universel de l’ADN dans le phage M13 permettant de transformer facilement et à volonté l’ADN double brin à séquencer en ADN monobrin analysable, ce qui a modifié totalement le paysage.14
Le génome de SV40 (1978)
De façon non surprenante, les premières retombées de la publication de ces deux méthodes de détermination de la structure primaire de l’ADN correspondirent au séquençage de plusieurs génomes viraux qui sont, avec les génomes bactériens, les plus petits génomes aisément accessibles. C’est ainsi qu’en 1978, soit à peine un an après la publication de Maxam et Gilbert, le groupe animé par Walter Fiers à Gand en Belgique publia dans la revue Nature la séquence complète soit 5 224 paires de bases du génome du virus simien SV40.15 L’importance de ce travail est grande par de nombreux aspects. Premièrement, il établissait de façon indépendante de leurs concepteurs l’efficacité de la méthode de séquençage par dégradation chimique de Maxam et Gilbert sur un génome de virus animal, dont la contamination des cultures des premiers vaccins contre la poliomyélite défraya la chronique. Par ailleurs en lien avec les circonstances de sa découverte, ce virus est responsable du développement de tumeurs in vitro chez le rat et à cet égard est l’objet de très nombreuses recherches. Sur le plan biologique, la détermination de cette séquence fournissait des bases solides pour les études ultérieures en établissant sans ambiguïté la structure des protéines codées par le génome, sur l’antigène T mais aussi sur celle de l’origine de la replication du génome viral. Il peut paraître surprenant que si peu de temps se soit écoulé entre la publication de Maxam et Gilbert et celle de ce génome viral. En réalité, Walter Fiers avait débuté ce séquençage bien avant en mettant à profit les méthodes de séquençage de l’ARN développées par le groupe de Cambridge pour séquencer nombre d’ARN messagers. Toutefois, la méthode de séquençage de l’ADN permit à son groupe de finaliser ce travail en un temps record.
Le virus de l’hépatite B (1979)
L’hépatite B est une maladie virale particulièrement fréquente, puisqu’on estime que 3 à 6 % de la population mondiale serait infectée et que jusqu’à un tiers de la population aurait été infectée. Si dans la majorité des cas, l’évolution de la maladie est très favorable, il faut noter l’existence de formes fulminantes mortelles et l’évolution vers la chronicité qui n’est pas rare. En 2005 on comptait, dans le monde, quelques 350 millions de porteurs chroniques, pour lesquels un risque élevé de cirrhose ou de cancer du foie existait. Le virus de l’hépatite B (HPV) a été découvert par Baruch S. Blumberg en 1963, travail pour lequel il fut honoré du prix Nobel de médecine ou physiologie en 1976.
Le génome de l’hépatite B a une structure particulière unique. C’est un ADN partiellement double brin, linéaire mais prenant une structure circulaire par hybridation de la petite chaîne sur la grande complétant la partie manquante de celle-ci. C’est Pierre Tiollais qui le premier m’évoqua le projet, hasardeux à l’époque, de déterminer la séquence nucléotidique de ce virus, projet doublement difficile dans la mesure où le virus n’infecte que l’homme et le chimpanzé, qu’il n’existe pas de système de culture cellulaire apte à le produire, et que les méthodes de séquençage de l’ADN venaient à peine d’être publiées. La première difficulté fut contournée par le clonage moléculaire du génome viral dans celui du phage lambda gtWES qui, après infection de la bactérie Escherichia coli C600recBC, permet de produire en masse l’ADN recombinant. Ce travail fut réalisé, du moins au début, en Suède dans le laboratoire de L. Philippsson car, à l’époque, la recombinaison in vitro était interdite en France.16
Ayant à disposition de l’ADN marqué au 32P, la séquence nucléotidique complète du génome de l’HBV fut déterminée par la méthode des dégradations chimiques de Maxam et Gilbert, mais également en parallèle par la méthode aux ddN. Ce travail montra que ce génome de 3 182 nucléotides après complétion des deux chaînes pour le clonage moléculaire présente quatre phases de lecture ouvertes codant des peptides de plus de 100 acides aminés dont celle de l’antigène de surface HBs. Toutes les quatre sont situées sur la grande chaîne. À ce jour aucune autre phase ouverte ne s’est avérée fonctionnelle.17
La protéine d’enveloppe codée par le gène S sera exprimée tout d’abord dans E coli, puis dans la levure pour devenir le premier vaccin recombinant. De façon ironique la France est le seul pays parmi les 177 pays pratiquant cette vaccination à connaître une controverse sur sa sécurité. Il n’en reste pas moins que ce vaccin fait partie des 10 vaccins obligatoires du nourrisson tout en étant recommandé chez l’adolescent.18
Le génome de l’hépatite B a une structure particulière unique. C’est un ADN partiellement double brin, linéaire mais prenant une structure circulaire par hybridation de la petite chaîne sur la grande complétant la partie manquante de celle-ci. C’est Pierre Tiollais qui le premier m’évoqua le projet, hasardeux à l’époque, de déterminer la séquence nucléotidique de ce virus, projet doublement difficile dans la mesure où le virus n’infecte que l’homme et le chimpanzé, qu’il n’existe pas de système de culture cellulaire apte à le produire, et que les méthodes de séquençage de l’ADN venaient à peine d’être publiées. La première difficulté fut contournée par le clonage moléculaire du génome viral dans celui du phage lambda gtWES qui, après infection de la bactérie Escherichia coli C600recBC, permet de produire en masse l’ADN recombinant. Ce travail fut réalisé, du moins au début, en Suède dans le laboratoire de L. Philippsson car, à l’époque, la recombinaison in vitro était interdite en France.16
Ayant à disposition de l’ADN marqué au 32P, la séquence nucléotidique complète du génome de l’HBV fut déterminée par la méthode des dégradations chimiques de Maxam et Gilbert, mais également en parallèle par la méthode aux ddN. Ce travail montra que ce génome de 3 182 nucléotides après complétion des deux chaînes pour le clonage moléculaire présente quatre phases de lecture ouvertes codant des peptides de plus de 100 acides aminés dont celle de l’antigène de surface HBs. Toutes les quatre sont situées sur la grande chaîne. À ce jour aucune autre phase ouverte ne s’est avérée fonctionnelle.17
La protéine d’enveloppe codée par le gène S sera exprimée tout d’abord dans E coli, puis dans la levure pour devenir le premier vaccin recombinant. De façon ironique la France est le seul pays parmi les 177 pays pratiquant cette vaccination à connaître une controverse sur sa sécurité. Il n’en reste pas moins que ce vaccin fait partie des 10 vaccins obligatoires du nourrisson tout en étant recommandé chez l’adolescent.18
Le génome mitochondrial (1981)
La mitochondrie, petit organite présent en très grand nombre dans toutes les cellules eucaryotes et dont le rôle est de fournir à la cellule hôte son ATP, possède un petit génome circulaire. Bien que la majorité des protéine mitochondriales soient codées par le génome nucléaire et importées après synthèse dans la mitochondrie, une partie des protéines mitochondriales est codée par le génome mitochondrial et synthétisée par une machinerie cellulaire propre à la mitochondrie. Il n’a pas fallu longtemps au groupe de Cambridge devant l’importance et les particularités biologiques de la mitochondrie alliées à la facilité de préparation de son ADN pour s’intéresser à celle-ci et se lancer dans l’analyse de sa structure primaire de son génome, publié en 1981.19 Il a une longueur de 16 569 paires de base avec une distribution de gènes extrêmement compacte confirmant l’utilisation d’un code génétique particulier codant, outre les éléments de sa propre machinerie de synthèse, des protéines de plusieurs gènes indispensables au rôle fondamental de la mitochondrie en tant que fournisseur d’énergie à sa cellule hôte.
La levure de boulanger (1992-1996)
Dans la liste forcément incomplète des génomes qui furent l’objet des premiers travaux de séquençage, une mention spéciale doit être faite pour celui de la levure Saccharomyces. En 1986, Renato Dulbecco publia dans la revue Science un article intitulé A turning point in cancer research: sequencing the human genome dans lequel il recommandait l’abandon de l’approche réductionniste suivie à l’époque par les biologistes pensant que l’analyse individuelle des gènes leurs permettrait de comprendre des phénomènes intégrés mettant en jeu de nombreux gènes au profit d’une méthode globale d’analyse du génome.20 Convaincu lui-même par les arguments développés dans cet article, André Goffeau convainquit à son tour Fernand Van Hoeck et Jean Dreux de Nettencourt, responsables du programme de biotechnologie des Communautés européennes, de l’intérêt que représenterait la connaissance complète du génome de la levure de boulangerie Saccharomyces cerevisiae. Les 16 chromosomes de la levure furent confiés à différents responsables, lesquels eurent en charge de coordonner le séquençage des cosmides dans lesquels était cloné le génome d’un chromosome donné en les distribuant à un petit nombre de laboratoires. En 1992 fut publiée la séquence du premier chromosome, séquence qui fut suivie par celle des 15 autres et, en 1996, sortit un article récapitulatif de ce travail décrivant l’intégralité du premier génome eucaryote. Cette séquence révélait une très forte compaction de l’information (peu d’introns, peu de pseudogènes). Elle se limitait à quelque 6 000 gènes dont 5 500 environ codaient des protéines. De façon inattendue, un tiers d’entre elles exercent des fonctions inconnues tandis que moins de 20 % sont strictement dévolues à la croissance. Enfin, on constata plus tard avec surprise qu’un grand nombre étaient homologues à des protéines humaines ou des plantes supérieures.21
Références
1. Lander E S, Lindon LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature 2001;409:860-921.
2. Venter JC, Adams MD, Myers EW, et al. The sequence of the human genome. Science 2001;291:1304-51.
3. Arber W, Dussoix D. Host specificity of DNA produced by Escherichia coli. I. Host controlled modification of bacteriophage lambda. J Mol Biol 1962;5:18-36.
4. Smith H0, Wilcox KW. A restriction enzyme from Haemophilus influenzae. I. Purification and general properties. J Mol Biol 1970;51:379-91.
5. Danna KJ, Nathans D. Specific cleavage of simian virus 40 DNA by restriction endonuclease of Hemophilus influenzae. Proc Natl Acad Sci (USA) 1971;68:2913-7.
6. Wu R, Kaiser AD. Structure and base sequence in the cohesive ends of bacteriophage lambda DNA. J Mol Biol 1968;35:523-37.
7. Ling V. Pyrimidine sequences from the DNA of bacteriophages fd, fl, and X174 . Proc Natl Acad Sci U S A 1972;69:742-6.
8. Ziff EB, Sedat JW, Galibert F. Determination of the nucleotide sequence of a fragment of bacteriophage phiX 174 DNA. Nat New Biol. 1973; 241(106):34-7.
9. Sanger F, Donelson JE, Coulson AR, Kössel H, Fischer D. Use of DNa polymerase 1 primed by a synthetiqc oligonucleotide to determine a nucleotide sequence in phage f1 DNA. Proc Natl Acad Sci U S A 1973;70:1209-13.
10. Sanger F, Air GM, Barrell BG, et al. Nucleotide sequence of bacteriophage phi X174 DNA. Nature 1977;265:687-95.
11. Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A 1977;74:5463-7.
12. Gilbert W, Maxam, A. The nucleotide sequence of the lac operator. Proc Natl Acad Sci USA 1973;70:3581-4
13. Maxam AM, Gilbert W. A new method for sequencing DNA. Proc Natl Acad Sci USA 1977;74:560-4.
14. Messing J, Gronenborn, B, Muller-Hill B, Hans Hofschneider P. FilamentouscoliphageM13 as a cloning vehicle: Insertion of a HindII fragment of the lac regulatory regionin M13 replicative form in vitro. Proc Natl Acad USA 1977;74:3642-6. W Arber
15. Fiers W, Contreras R, Haegemann G, et al. Complete nucleotide sequence of SV40 DNA. Nature 1978;273:113-20.
16. Fritsch A, Pourcel C, Charnay P, Tiolais P. Cloning of the hepatitis B virus genome in Escherichia coli. CR Hebd Seances Acad Sci D 1978;287:1453-6.
17. Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature 1979;281:646-50.
18. Charnay P, Gervais M, Louise A, Galibert F, Tiollais P. Biosynthesis of hepatitis B virus surface antigen in Escherichia coli. Nature 1980;286:893-5.
19. Anderson S, Bankier AT, Barrell BG, et al. Sequence and organization of the human mitochondrial genome. Nature 1981;290:457-65.
20. Dulbecco R. A turning point in cancer research: sequencing the human genome. Science 1986;231:1055-6.
21. Goffeau A, Barrell BG, Bussey H, et al. Life with 6000 genes. Science 1996;274:546-67.
2. Venter JC, Adams MD, Myers EW, et al. The sequence of the human genome. Science 2001;291:1304-51.
3. Arber W, Dussoix D. Host specificity of DNA produced by Escherichia coli. I. Host controlled modification of bacteriophage lambda. J Mol Biol 1962;5:18-36.
4. Smith H0, Wilcox KW. A restriction enzyme from Haemophilus influenzae. I. Purification and general properties. J Mol Biol 1970;51:379-91.
5. Danna KJ, Nathans D. Specific cleavage of simian virus 40 DNA by restriction endonuclease of Hemophilus influenzae. Proc Natl Acad Sci (USA) 1971;68:2913-7.
6. Wu R, Kaiser AD. Structure and base sequence in the cohesive ends of bacteriophage lambda DNA. J Mol Biol 1968;35:523-37.
7. Ling V. Pyrimidine sequences from the DNA of bacteriophages fd, fl, and X174 . Proc Natl Acad Sci U S A 1972;69:742-6.
8. Ziff EB, Sedat JW, Galibert F. Determination of the nucleotide sequence of a fragment of bacteriophage phiX 174 DNA. Nat New Biol. 1973; 241(106):34-7.
9. Sanger F, Donelson JE, Coulson AR, Kössel H, Fischer D. Use of DNa polymerase 1 primed by a synthetiqc oligonucleotide to determine a nucleotide sequence in phage f1 DNA. Proc Natl Acad Sci U S A 1973;70:1209-13.
10. Sanger F, Air GM, Barrell BG, et al. Nucleotide sequence of bacteriophage phi X174 DNA. Nature 1977;265:687-95.
11. Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci U S A 1977;74:5463-7.
12. Gilbert W, Maxam, A. The nucleotide sequence of the lac operator. Proc Natl Acad Sci USA 1973;70:3581-4
13. Maxam AM, Gilbert W. A new method for sequencing DNA. Proc Natl Acad Sci USA 1977;74:560-4.
14. Messing J, Gronenborn, B, Muller-Hill B, Hans Hofschneider P. FilamentouscoliphageM13 as a cloning vehicle: Insertion of a HindII fragment of the lac regulatory regionin M13 replicative form in vitro. Proc Natl Acad USA 1977;74:3642-6. W Arber
15. Fiers W, Contreras R, Haegemann G, et al. Complete nucleotide sequence of SV40 DNA. Nature 1978;273:113-20.
16. Fritsch A, Pourcel C, Charnay P, Tiolais P. Cloning of the hepatitis B virus genome in Escherichia coli. CR Hebd Seances Acad Sci D 1978;287:1453-6.
17. Galibert F, Mandart E, Fitoussi F, Tiollais P, Charnay P. Nucleotide sequence of the hepatitis B virus genome (subtype ayw) cloned in E. coli. Nature 1979;281:646-50.
18. Charnay P, Gervais M, Louise A, Galibert F, Tiollais P. Biosynthesis of hepatitis B virus surface antigen in Escherichia coli. Nature 1980;286:893-5.
19. Anderson S, Bankier AT, Barrell BG, et al. Sequence and organization of the human mitochondrial genome. Nature 1981;290:457-65.
20. Dulbecco R. A turning point in cancer research: sequencing the human genome. Science 1986;231:1055-6.
21. Goffeau A, Barrell BG, Bussey H, et al. Life with 6000 genes. Science 1996;274:546-67.