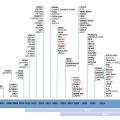
Les maladies auto-inflammatoires (MAI) représentent un modèle captivant de pathologies ayant bénéficié des outils modernes de la génétique, notamment le séquençage en haut débit ou NGS (next generation sequencing).
Multiples modes de transmission héréditaires
Ces approches ont progressivement mis en lumière la complexité d’un groupe de maladies que l’on pensait de transmission simple, monogénique autosomique pour les premières fièvres récurrentes héréditaires (FRH) identifiées, et multifactorielle pour certaines autres.1 De nouveaux modes de transmission héréditaires ont été révélés ces dernières années, non seulement pour les gènes découverts récemment mais également pour des gènes connus depuis vingt-cinq ans comme le gène MEFV (MEditerranean FeVer) classiquement responsable de la fièvre méditerranéenne (FMF)2,3 ou le gène MVK (mévalonate kinase), responsable du déficit en mévalonate kinase (MKD).4,5
Une centaine de MAI monogéniques identifiées
Chacune de ces maladies est rare individuellement (sauf la FMF dans les populations à risque), mais la liste des gènes impliqués s’allonge au fur et à mesure des connaissances, on en compte plus de 100 à ce jour. Un tableau des formes cliniques de MAI, les gènes en cause et les modes de transmission correspondants est disponible sur Infevers, site dédié aux mutations des MAI (https://infevers.umai-montpellier.fr/Classification_AID/page1.html).6 Quelques exemples représentatifs sont détaillés ci-dessous.
figure ). Deux de ces FRH sont classiquement récessives (FMF et MKD), mais des formes cliniques différentes sont associées à une hérédité dominante.
La FMF est une maladie autosomique récessive dont quatre principales mutations dans l’exon 10 du gène MEFV se sont répandues dans les pays méditerranéens par effet fondateur en raison d’endogamie culturelle dans les populations à risque (arméniennes, turques, juives séfarades et maghrébines).7 On sait maintenant que 20 à 40 % des patients avec des critères cliniques de FMF sont hétérozygotes, bien que le mécanisme sous-jacent ne soit pas encore totalement élucidé.8-11 La présence d’une mutation exonique rare ou intronique, non capturée par la technique de diagnostic utilisée, ainsi qu’une régulation épigénétique ont été évoquées. Certaines formes de « FMF-like » dominantes sont liées à des mutations dans les codons 373 dans l’exon 3,12 478 dans l’exon 5,13 577 et 583 dans l’exon 8,14 694 dans l’exon 10.15
Plus intriguant, un nouveau syndrome appelé pyrin-associated autoinflammation with neutrophilic dermatosis (PAAND), caractérisé par une fièvre prolongée et une dermatose neutrophilique, a été décrit en 2016 comme étant dû à une mutation en position 242 ou 244 de la protéine codée par MEFV, la pyrine.16 Lorsque celle-ci est phosphorylée par les sérine-thréonine kinases PKN1 et PKN2 aux résidus p.S208 et p.S242, la pyrine se lie à la protéine 14-3-3, ce qui la maintient dans son état inactif. Les modèles in vitro montrent que la mutation p.S242R de la pyrine est constitutivement déphosphorylée, avec une réduction de la liaison 14-3-3. L’augmentation de l’activité de l’inflammasome de la pyrine qui en résulte et la production accrue d’IL-1β semblent être à l’origine de la pathologie dans le PAAND. Il est donc très important pour le diagnostic de prendre en compte conjointement clinique, nombre et localisation des variants pathogènes.
Le MKD est aussi typiquement une maladie de transmission autosomique récessive qui présente un éventail clinique systémique large, allant de la fièvre récurrente anciennement (mal) nommée hyper-IgD dans sa forme modérée, jusqu’à une forme métabolique grave (déficit complet ou acidurie mévalonique).4,5 Des anomalies génétiques dans ce même gène MVK ont curieusement été associées aussi à une maladie purement dermatologique, la porokératose, dont le mode de transmission est dominant.17 Dans la forme systémique MKD, deux mutations sont héritées de chaque parent (bi-allélisme). Dans la forme de porokératose cutanée localisée, les deux mutations (ou plus) se produisent de manière séquentielle : la première est germinale et héritée de manière dominante d’un parent, et la seconde est acquise plus tard dans la vie, sur l’allèle de type sauvage, probablement après une altération par les UVA de l’ADN des cellules de l’épiderme dans les zones de peau exposées au soleil pour certains des variants pathogènes.18
Les deux autres FRH, TNF receptor-associated periodic syndrome (TRAPS) et cryopyrin-associated periodic syndrome (CAPS), sont transmises sur le mode dominant et sont dues à des mutations hétérozygotes dans les gènes TNFRSF1A et NLRP3 respectivement.19,20 Alors qu’aucune mutation n’avait été identifiée par la technique gold standard de l’époque – le séquençage Sanger – chez des patients cliniquement typiques, une réanalyse des gènes par NGS a permis de découvrir des mutations de novo en mosaïque faible dans les gènes concernés.21
La première est une MAI systémique liée à l’X (NEMO-NDAS) associée à des mutations de novo dans le gène IKBKG, codant la protéine NEMO.22,23 Elle apparaît dès les premiers mois de vie et associe adénopathies, hépatosplénomégalie, fièvre, panniculite, éruption cutanée nodulaire et signature de l’interféron de type I. Toutes les mutations connues à ce jour sont prédites pour altérer l’épissage et induire une délétion de l’exon 5, ce qui la différencie des autres maladies monogéniques associées à ce même gène comme l’incontinentia pigmenti et la dysplasie ectodermique.22
La même année, l’identification du syndrome VEXAS (vacuoles, E1 enzyme, X-linked, autoinflammatory, somatic) a fait grand bruit. Il s’agit d’une MAI causée par une mutation somatique dans le gène UBA1 expliquant sa survenue tardive, autour de l’âge de 60 ans.24
Transmissions autosomiques, le plus souvent récessives
Les quatre premiers gènes de FRH ont été identifiés entre 1997 et 2001 (La FMF est une maladie autosomique récessive dont quatre principales mutations dans l’exon 10 du gène MEFV se sont répandues dans les pays méditerranéens par effet fondateur en raison d’endogamie culturelle dans les populations à risque (arméniennes, turques, juives séfarades et maghrébines).7 On sait maintenant que 20 à 40 % des patients avec des critères cliniques de FMF sont hétérozygotes, bien que le mécanisme sous-jacent ne soit pas encore totalement élucidé.8-11 La présence d’une mutation exonique rare ou intronique, non capturée par la technique de diagnostic utilisée, ainsi qu’une régulation épigénétique ont été évoquées. Certaines formes de « FMF-like » dominantes sont liées à des mutations dans les codons 373 dans l’exon 3,12 478 dans l’exon 5,13 577 et 583 dans l’exon 8,14 694 dans l’exon 10.15
Plus intriguant, un nouveau syndrome appelé pyrin-associated autoinflammation with neutrophilic dermatosis (PAAND), caractérisé par une fièvre prolongée et une dermatose neutrophilique, a été décrit en 2016 comme étant dû à une mutation en position 242 ou 244 de la protéine codée par MEFV, la pyrine.16 Lorsque celle-ci est phosphorylée par les sérine-thréonine kinases PKN1 et PKN2 aux résidus p.S208 et p.S242, la pyrine se lie à la protéine 14-3-3, ce qui la maintient dans son état inactif. Les modèles in vitro montrent que la mutation p.S242R de la pyrine est constitutivement déphosphorylée, avec une réduction de la liaison 14-3-3. L’augmentation de l’activité de l’inflammasome de la pyrine qui en résulte et la production accrue d’IL-1β semblent être à l’origine de la pathologie dans le PAAND. Il est donc très important pour le diagnostic de prendre en compte conjointement clinique, nombre et localisation des variants pathogènes.
Le MKD est aussi typiquement une maladie de transmission autosomique récessive qui présente un éventail clinique systémique large, allant de la fièvre récurrente anciennement (mal) nommée hyper-IgD dans sa forme modérée, jusqu’à une forme métabolique grave (déficit complet ou acidurie mévalonique).4,5 Des anomalies génétiques dans ce même gène MVK ont curieusement été associées aussi à une maladie purement dermatologique, la porokératose, dont le mode de transmission est dominant.17 Dans la forme systémique MKD, deux mutations sont héritées de chaque parent (bi-allélisme). Dans la forme de porokératose cutanée localisée, les deux mutations (ou plus) se produisent de manière séquentielle : la première est germinale et héritée de manière dominante d’un parent, et la seconde est acquise plus tard dans la vie, sur l’allèle de type sauvage, probablement après une altération par les UVA de l’ADN des cellules de l’épiderme dans les zones de peau exposées au soleil pour certains des variants pathogènes.18
Les deux autres FRH, TNF receptor-associated periodic syndrome (TRAPS) et cryopyrin-associated periodic syndrome (CAPS), sont transmises sur le mode dominant et sont dues à des mutations hétérozygotes dans les gènes TNFRSF1A et NLRP3 respectivement.19,20 Alors qu’aucune mutation n’avait été identifiée par la technique gold standard de l’époque – le séquençage Sanger – chez des patients cliniquement typiques, une réanalyse des gènes par NGS a permis de découvrir des mutations de novo en mosaïque faible dans les gènes concernés.21
Mutations liées à l’X de découverte récente
Aucune MAI liée à l’X n’était connue jusqu’à la découverte, en 2020, de deux nouveaux gènes localisés sur ce chromosome, dont les mutations causent une MAI n’apparaissant quasi exclusivement que chez les hommes, par essence « hémizygotes ».La première est une MAI systémique liée à l’X (NEMO-NDAS) associée à des mutations de novo dans le gène IKBKG, codant la protéine NEMO.22,23 Elle apparaît dès les premiers mois de vie et associe adénopathies, hépatosplénomégalie, fièvre, panniculite, éruption cutanée nodulaire et signature de l’interféron de type I. Toutes les mutations connues à ce jour sont prédites pour altérer l’épissage et induire une délétion de l’exon 5, ce qui la différencie des autres maladies monogéniques associées à ce même gène comme l’incontinentia pigmenti et la dysplasie ectodermique.22
La même année, l’identification du syndrome VEXAS (vacuoles, E1 enzyme, X-linked, autoinflammatory, somatic) a fait grand bruit. Il s’agit d’une MAI causée par une mutation somatique dans le gène UBA1 expliquant sa survenue tardive, autour de l’âge de 60 ans.24
MAI oligogéniques, un mode de transmission qui complique le conseil génétique
L’oligogénisme est l’implication obligatoire de plus d’un gène pour que se déclenche la maladie. Ces gènes participent en général à la même voie de signalisation ou codent les différentes sous-unités d’une protéine multimérique.
Un mécanisme de digénisme a été prouvé pour la première fois dans les MAI pour un groupe d’interféronopathies appelées proteasome-associated autoinflammatory syndromes (PRAAS).25,26 Le premier syndrome PRAAS dû à des mutations autosomiques récessives (homozygotes ou hétérozygotes composites) dans le gène PSMB8 a été décrit en 2011.25,26 Quatre ans plus tard, Brehm et al. rapportaient le cas d’un patient avec une mutation dominante dans POMP, ainsi que ceux de six patients doubles hétérozygotes, c’est-à-dire présentant des variants pathogènes coexistant dans deux gènes : PSMB8 et PSMA3, PSMB8 et PSMB4, PSMB4 et PSMB9. Ils établissaient ainsi deux nouveaux modèles d’hérédité de PRAAS, autosomique dominant et digénique.27 Ce mode de transmission différentiel pour chaque gène complique le conseil génétique.
Un mécanisme de digénisme a été prouvé pour la première fois dans les MAI pour un groupe d’interféronopathies appelées proteasome-associated autoinflammatory syndromes (PRAAS).25,26 Le premier syndrome PRAAS dû à des mutations autosomiques récessives (homozygotes ou hétérozygotes composites) dans le gène PSMB8 a été décrit en 2011.25,26 Quatre ans plus tard, Brehm et al. rapportaient le cas d’un patient avec une mutation dominante dans POMP, ainsi que ceux de six patients doubles hétérozygotes, c’est-à-dire présentant des variants pathogènes coexistant dans deux gènes : PSMB8 et PSMA3, PSMB8 et PSMB4, PSMB4 et PSMB9. Ils établissaient ainsi deux nouveaux modèles d’hérédité de PRAAS, autosomique dominant et digénique.27 Ce mode de transmission différentiel pour chaque gène complique le conseil génétique.
MAI multifactorielles avec une implication de l’environnement
Contrairement aux formes mono- ou oligogéniques, les MAI multifactorielles sont moins nombreuses, mais le nombre de patients pour chacune d’elles est plus grand.
Dans ce type d’hérédité, l’implication de l’environnement est grande même si elle n’est pas toujours bien comprise, et celle des centaines ou milliers de gènes est individuellement infime et donc insuffisante pour déclencher la maladie, laquelle apparaît généralement à l’âge adulte (par exemple, dans la maladie de Crohn, la maladie de Behçet ou l’arthrite juvénile idiopathique). Chez les adultes, ces formes ne sont généralement pas éligibles au diagnostic génétique.
En pédiatrie, il faut néanmoins garder en tête la possibilité de formes monogéniques vraies chez les patients très jeunes, comme les MAI à type de maladies inflammatoires chroniques de l’intestin (MICI) liées à des mutations dans les gènes de la voie de l’IL-1028 ou dans le gène ADAM17 ;29 un « Behçet » pédiatrique pouvant cacher une haplo-insuffisance en A20 (HA20) due à des mutations dominantes dans le gène TNFAIP330 ou un MKD ;31 ou une arthrite juvénile systémique due à des mutations récessives dans le gène LACC1.32
Dans ce type d’hérédité, l’implication de l’environnement est grande même si elle n’est pas toujours bien comprise, et celle des centaines ou milliers de gènes est individuellement infime et donc insuffisante pour déclencher la maladie, laquelle apparaît généralement à l’âge adulte (par exemple, dans la maladie de Crohn, la maladie de Behçet ou l’arthrite juvénile idiopathique). Chez les adultes, ces formes ne sont généralement pas éligibles au diagnostic génétique.
En pédiatrie, il faut néanmoins garder en tête la possibilité de formes monogéniques vraies chez les patients très jeunes, comme les MAI à type de maladies inflammatoires chroniques de l’intestin (MICI) liées à des mutations dans les gènes de la voie de l’IL-1028 ou dans le gène ADAM17 ;29 un « Behçet » pédiatrique pouvant cacher une haplo-insuffisance en A20 (HA20) due à des mutations dominantes dans le gène TNFAIP330 ou un MKD ;31 ou une arthrite juvénile systémique due à des mutations récessives dans le gène LACC1.32
Génétique des MAI en pratique
Quand demander un test génétique ?
L’indication majeure d’un test génétique est la suspicion d’une forme mono- ou digénique de MAI. Certains éléments peuvent orienter :
– l’âge de début précoce des symptômes de la maladie. Cependant, des néomutations somatiques à l’origine de mosaïques tissulaires comme dans les CAPS ou plus rarement les TRAPS peuvent survenir, dans de rares cas, à l’âge adulte.21
– le caractère familial de l’affection, notion majeure à rechercher à l’interrogatoire et qui n’est pas suffisamment renseignée ou connue. Néanmoins, l’absence de cas multiples dans la famille ne doit pas exclure une MAI héréditaire, car les cas apparaissent de façon sporadique dans près de trois quarts des cas de maladies récessives et dans les MAI dominantes à néomutations ;
– l’origine ethnique « méditerranéenne » en cas de suspicion de FMF ;
– la positivité d’un test fonctionnel (acidurie mévalonique, signature interféron, activité enzymatique ADA2 déficitaire, par exemple).
Un arbre décisionnel est proposé par un groupe d’experts de la Société internationale des MAI (ISSAID).33 En cas de doute, il est utile de présenter le dossier du patient lors d’une réunion de concertation pluridisciplinaire (RCP) spécialisée (Centre de référence des maladies auto-inflammatoires et des amyloses [CeRéMAIA] ou Filière de santé des maladies auto-immunes et auto-inflammatoires rares [FAI2R] ; informations sur www.ceremaia.fr).
– l’âge de début précoce des symptômes de la maladie. Cependant, des néomutations somatiques à l’origine de mosaïques tissulaires comme dans les CAPS ou plus rarement les TRAPS peuvent survenir, dans de rares cas, à l’âge adulte.21
– le caractère familial de l’affection, notion majeure à rechercher à l’interrogatoire et qui n’est pas suffisamment renseignée ou connue. Néanmoins, l’absence de cas multiples dans la famille ne doit pas exclure une MAI héréditaire, car les cas apparaissent de façon sporadique dans près de trois quarts des cas de maladies récessives et dans les MAI dominantes à néomutations ;
– l’origine ethnique « méditerranéenne » en cas de suspicion de FMF ;
– la positivité d’un test fonctionnel (acidurie mévalonique, signature interféron, activité enzymatique ADA2 déficitaire, par exemple).
Un arbre décisionnel est proposé par un groupe d’experts de la Société internationale des MAI (ISSAID).33 En cas de doute, il est utile de présenter le dossier du patient lors d’une réunion de concertation pluridisciplinaire (RCP) spécialisée (Centre de référence des maladies auto-inflammatoires et des amyloses [CeRéMAIA] ou Filière de santé des maladies auto-immunes et auto-inflammatoires rares [FAI2R] ; informations sur www.ceremaia.fr).
Quel séquençage ?
Il n’y a pas d’homogénéité dans l’offre de tests en France ni à l’étranger. Orphanet (www.orpha.net) liste les laboratoires spécialisés en fonction de la maladie suspectée.
Le généticien moléculaire décide de la technique qu’il souhaite utiliser en fonction de l’intérêt médical et de son organisation interne et peut en discuter avec le prescripteur.
Le séquençage classique par la technique de Sanger diminue au profit du haut débit, car il ne permet l’analyse que d’un (ou d’un petit nombre) d’exon(s).
Le séquençage NGS a plusieurs niveaux de complexité : ciblé sur quelques gènes responsables de MAI (panels) plus ou moins soumis à prérequis ou RCP, ou non ciblé et dirigé sur tout l’exome (c’est-à-dire toutes les séquences codant des protéines) ou tout le génome (whole genome sequencing [WGS]). Ces deux dernières approches sont obligatoirement soumises à l’accord d’une RCP, car la frontière entre le diagnostic et la recherche est floue et elles sont sujettes à l’identification fortuite de données incidentes (des variants pathogènes portés par le patient ou ses apparentés mais qui sont sans relation avec l’indication du test génétique). Les indications sont résumées dans un tableau que nous avons mis à disposition sur le site du laboratoire de Montpellier : https://umai-montpellier.fr/mai.php?display=1.
Le WGS peut se faire sur les deux plateformes nationales développées par le ministère de la Santé dans le cadre du Plan France médecine génomique 2025. L’une, Seqoia (https://laboratoire-seqoia.fr/) à Paris, est disponible pour les patients du nord de la France. L’autre, Auragen (https://www.auragen.fr/), en Auvergne-Rhône-Alpes, est destinée aux autres patients.
Le généticien moléculaire décide de la technique qu’il souhaite utiliser en fonction de l’intérêt médical et de son organisation interne et peut en discuter avec le prescripteur.
Le séquençage classique par la technique de Sanger diminue au profit du haut débit, car il ne permet l’analyse que d’un (ou d’un petit nombre) d’exon(s).
Le séquençage NGS a plusieurs niveaux de complexité : ciblé sur quelques gènes responsables de MAI (panels) plus ou moins soumis à prérequis ou RCP, ou non ciblé et dirigé sur tout l’exome (c’est-à-dire toutes les séquences codant des protéines) ou tout le génome (whole genome sequencing [WGS]). Ces deux dernières approches sont obligatoirement soumises à l’accord d’une RCP, car la frontière entre le diagnostic et la recherche est floue et elles sont sujettes à l’identification fortuite de données incidentes (des variants pathogènes portés par le patient ou ses apparentés mais qui sont sans relation avec l’indication du test génétique). Les indications sont résumées dans un tableau que nous avons mis à disposition sur le site du laboratoire de Montpellier : https://umai-montpellier.fr/mai.php?display=1.
Le WGS peut se faire sur les deux plateformes nationales développées par le ministère de la Santé dans le cadre du Plan France médecine génomique 2025. L’une, Seqoia (https://laboratoire-seqoia.fr/) à Paris, est disponible pour les patients du nord de la France. L’autre, Auragen (https://www.auragen.fr/), en Auvergne-Rhône-Alpes, est destinée aux autres patients.
Pratique de la génétique complexe
La pratique de la génétique des MAI se complexifie de jour en jour, mais les liens entre cliniciens et biologistes se resserrent en parallèle sous la forme de RCP, qu’il ne faut pas hésiter à solliciter, permettant de diminuer l’errance diagnostique. L’impasse diagnostique devrait aussi diminuer grâce à la généralisation du WGS.
Références
1. Sarrabay G, Barat-Houari M, Annakib S, Touitou I. The autoinflammatory diseases: A fashion with blurred boundaries! Semin Immunopathol 2015;37(4):359‑62.
2. French FMF Consortium. A candidate gene for familial mediterranean fever. Nat Genet 1997;17(1):25‑31.
3. The International FMF Consortium. Ancient missense mutations in a new member of the RoRet gene family are likely to cause familial mediterranean fever. Cell 1997;90(4):797‑807.
4. Drenth JP, Cuisset L, Grateau G, Vasseur C, van de Velde-Visser SD, de Jong JG, et al. Mutations in the gene encoding mevalonate kinase cause hyper-IgD and periodic fever syndrome. International Hyper-IgD Study Group. Nat Genet 1999;22(2):178‑81.
5. Houten SM, Kuis W, Duran M, de Koning TJ, van Royen-Kerkhof A, Romeijn GJ, et al. Mutations in MVK, encoding mevalonate kinase, cause hyperimmunoglobulinaemia D nd periodic fever syndrome. Nat Genet 1999;22(2):175‑7.
6. Milhavet F, Cuisset L, Hoffman HM, Slim R, El-Shanti H, Aksentijevich I, et al. The infevers autoinflammatory mutation online registry: Update with new genes and functions. Hum Mutat 2008;29(6):803‑8.
7. Shodat M, Halpern GJ. Familial mediterranean fever: A review. Genet Med 2011;13(6):487-98.
8. Güngörer V, Yorulmaz A, Arslan Ş. The clinical effect of gene mutations in familial Mediterranean fever disease. Pediatr Int 2021;64(1):e15052.
9. Beshlawy AE, Zekri AER, Ramadan MS, Selim YMM, Abdel-Salam A, Hegazy MT, et al. Genotype-phenotype associations in familial Mediterranean fever: A study of 500 Egyptian pediatric patients. Clin Rheumatol 2022;41(5):1511‑21.
10. Yildirim D, Bakkaloğlu SA, Soysal Acar AŞ, Çelik B, Buyan N. Evaluation of caregiver burden and coping strategies in parents of paediatric familial mediterranean fever patients in relation to illness severity, therapy and health-related quality of life. Qual Life Res 2021;30(5):1347‑54.
11. Alzyoud R, Alsweiti M, Maittah H, Zreqat E, Alwahadneh A, Abu-Shukair M, et al. Familial mediterranean fever in jordanian children: Single centre experience. Mediterr J Rheumatol 2018;29(4):211‑6.
12. Rowczenio DM, Youngstein T, Trojer H, Omoyinmi E, Baginska A, Brogan P, et al. British kindred with dominant FMF associated with high incidence of AA amyloidosis caused by novel MEFV variant, and a review of the literature. Rheumatology (Oxford) 2020;59(3):554‑8.
13. Aldea A, Campistol JM, Arostegui JI, Rius J, Maso M, Vives J, et al. A severe autosomal-dominant periodic inflammatory disorder with renal AA amyloidosis and colchicine resistance associated to the MEFV H478Y variant in a spanish kindred: An unusual familial Mediterranean fever phenotype or another MEFV-associated periodic inflammatory disorder? Am J Med Genet A 2004;124A(1):67‑73.
14. Stoffels M, Szperl A, Simon A, Netea MG, Plantinga TS, van Deuren M, et al. MEFV mutations affecting pyrin amino acid 577 cause autosomal dominant autoinflammatory disease. Ann Rheum Dis 2014;73(2):455‑61. Wang Q, Jin T, Jian S, Han X, Song H, Zhou Q, et al. A dominant pathogenic MEFV mutation causes atypical pyrin-associated periodic syndromes. JCI Insight 2023;e172975.
15. Rowczenio DM, Iancu DS, Trojer H, Gilbertson JA, Gillmore JD, Wechalekar AD, et al. Autosomal dominant familial mediterranean fever in Northern European Caucasians associated with deletion of p.M694 residue-a case series and genetic exploration. Rheumatology (Oxford) 2017;56(2):209‑13.
16. Moghaddas F, Llamas R, De Nardo D, Martinez-Banaclocha H, Martinez-Garcia JJ, Mesa-Del-Castillo P, et al. A novel pyrin-associated autoinflammation with neutrophilic dermatosis mutation further defines 14-3-3 binding of pyrin and distinction to familial mediterranean Fever. Ann Rheum Dis 2017;76(12):2085‑94.
17. Zhang SQ, Jiang T, Li M, Zhang X, Ren YQ, Wei SC, et al. Exome sequencing identifies MVK mutations in disseminated superficial actinic porokeratosis. Nat Genet 2012;44(10):1156‑60.
18. Touitou I. Twists and turns of the genetic story of mevalonate kinase-associated diseases: A review. Genes Dis 2022;9(4):1000‑7.
19. Hoffman HM, Mueller JL, Broide DH, Wanderer AA, Kolodner RD. Mutation of a new gene encoding a putative pyrin-like protein causes familial cold autoinflammatory syndrome and Muckle-Wells syndrome. Nat Genet 2001;29(3):301‑5.
20. McDermott MF, Aksentijevich I, Galon J, McDermott EM, Ogunkolade BW, Centola M, et al. Germline mutations in the extracellular domains of the 55 kDa TNF receptor, TNFR1, define a family of dominantly inherited autoinflammatory syndromes. Cell 1999;97(1):133‑44.
21. Labrousse M, Kevorkian-Verguet C, Boursier G, Rowczenio D, Maurier F, Lazaro E, et al. Mosaicism in autoinflammatory diseases: Cryopyrin-associated periodic syndromes (CAPS) and beyond. A systematic review. Crit Rev Clin Lab Sci 2018;55(6):432‑42.
22. de Jesus AA, Hou Y, Brooks S, Malle L, Biancotto A, Huang Y, et al. Distinct interferon signatures and cytokine patterns define additional systemic autoinflammatory diseases. J Clin Invest 2020;130(4):1669‑82.
23. Lee Y, Wessel AW, Xu J, Reinke JG, Lee E, Kim SM, et al. Genetically programmed alternative splicing of NEMO mediates an autoinflammatory disease phenotype. J Clin Invest 2022;132(6):e128808.
24. Beck DB, Ferrada MA, Sikora KA, Ombrello AK, Collins JC, Pei W, et al. Somatic mutations in UBA1 and severe adult-onset autoinflammatory disease. N Engl J Med 2020;383(27):2628‑38.
25. Agarwal AK, Xing C, DeMartino GN, Mizrachi D, Hernandez MD, Sousa AB, et al. PSMB8 encoding the β5i proteasome subunit is mutated in joint contractures, muscle atrophy, microcytic anemia, and panniculitis-induced lipodystrophy syndrome. Am J Hum Genet 2010;87(6):866‑72.
26. Arima K, Kinoshita A, Mishima H, Kanazawa N, Kaneko T, Mizushima T, et al. Proteasome assembly defect due to a proteasome subunit beta type 8 (PSMB8) mutation causes the autoinflammatory disorder, Nakajo-Nishimura syndrome. Proc Natl Acad Sci USA 2011;108(36):14914‑9.
27. Brehm A, Liu Y, Sheikh A, Marrero B, Omoyinmi E, Zhou Q, et al. Additive loss-of-function proteasome subunit mutations in CANDLE/PRAAS patients promote type I IFN production. J Clin Invest 2015;125(11):4196‑211.
28. Krawiec P, Pawłowska-Kamieniak A, Pac-Kożuchowska E. Interleukin 10 and interleukin 10 receptor in paediatric inflammatory bowel disease: From bench to bedside lesson. J Inflamm (Lond) 2021;18(1):13.
29. Blaydon DC, Biancheri P, Di WL, Plagnol V, Cabral RM, Brooke MA, et al. Inflammatory skin and bowel disease linked to ADAM17 deletion. N Engl J Med 2011;365(16):1502‑8.
30. Zhou Q, Wang H, Schwartz DM, Stoffels M, Park YH, Zhang Y, et al. Loss-of-function mutations in TNFAIP3 leading to A20 haploinsufficiency cause an early-onset autoinflammatory disease. Nat Genet 2016;48(1):67‑73.
31. Koné-Paut I, Sanchez E, Le Quellec A, Manna R, Touitou I. Autoinflammatory gene mutations in Behçet’s disease. Ann Rheum Dis 2007;66(6):832‑4.
32. Wakil SM, Monies DM, Abouelhoda M, Al-Tassan N, Al-Dusery H, Naim EA, et al. Association of a mutation in LACC1 with a monogenic form of systemic juvenile idiopathic arthritis. Arthritis Rheumatol 2015;67(1):288‑95.
33. Shinar Y, Ceccherini I, Rowczenio D, Aksentijevich I, Arostegui J, Ben-Chétrit E, et al. ISSAID/EMQN best practice guidelines for the genetic diagnosis of monogenic autoinflammatory diseases in the next-generation sequencing era. Clin Chem 2020;66(4):525‑36.
2. French FMF Consortium. A candidate gene for familial mediterranean fever. Nat Genet 1997;17(1):25‑31.
3. The International FMF Consortium. Ancient missense mutations in a new member of the RoRet gene family are likely to cause familial mediterranean fever. Cell 1997;90(4):797‑807.
4. Drenth JP, Cuisset L, Grateau G, Vasseur C, van de Velde-Visser SD, de Jong JG, et al. Mutations in the gene encoding mevalonate kinase cause hyper-IgD and periodic fever syndrome. International Hyper-IgD Study Group. Nat Genet 1999;22(2):178‑81.
5. Houten SM, Kuis W, Duran M, de Koning TJ, van Royen-Kerkhof A, Romeijn GJ, et al. Mutations in MVK, encoding mevalonate kinase, cause hyperimmunoglobulinaemia D nd periodic fever syndrome. Nat Genet 1999;22(2):175‑7.
6. Milhavet F, Cuisset L, Hoffman HM, Slim R, El-Shanti H, Aksentijevich I, et al. The infevers autoinflammatory mutation online registry: Update with new genes and functions. Hum Mutat 2008;29(6):803‑8.
7. Shodat M, Halpern GJ. Familial mediterranean fever: A review. Genet Med 2011;13(6):487-98.
8. Güngörer V, Yorulmaz A, Arslan Ş. The clinical effect of gene mutations in familial Mediterranean fever disease. Pediatr Int 2021;64(1):e15052.
9. Beshlawy AE, Zekri AER, Ramadan MS, Selim YMM, Abdel-Salam A, Hegazy MT, et al. Genotype-phenotype associations in familial Mediterranean fever: A study of 500 Egyptian pediatric patients. Clin Rheumatol 2022;41(5):1511‑21.
10. Yildirim D, Bakkaloğlu SA, Soysal Acar AŞ, Çelik B, Buyan N. Evaluation of caregiver burden and coping strategies in parents of paediatric familial mediterranean fever patients in relation to illness severity, therapy and health-related quality of life. Qual Life Res 2021;30(5):1347‑54.
11. Alzyoud R, Alsweiti M, Maittah H, Zreqat E, Alwahadneh A, Abu-Shukair M, et al. Familial mediterranean fever in jordanian children: Single centre experience. Mediterr J Rheumatol 2018;29(4):211‑6.
12. Rowczenio DM, Youngstein T, Trojer H, Omoyinmi E, Baginska A, Brogan P, et al. British kindred with dominant FMF associated with high incidence of AA amyloidosis caused by novel MEFV variant, and a review of the literature. Rheumatology (Oxford) 2020;59(3):554‑8.
13. Aldea A, Campistol JM, Arostegui JI, Rius J, Maso M, Vives J, et al. A severe autosomal-dominant periodic inflammatory disorder with renal AA amyloidosis and colchicine resistance associated to the MEFV H478Y variant in a spanish kindred: An unusual familial Mediterranean fever phenotype or another MEFV-associated periodic inflammatory disorder? Am J Med Genet A 2004;124A(1):67‑73.
14. Stoffels M, Szperl A, Simon A, Netea MG, Plantinga TS, van Deuren M, et al. MEFV mutations affecting pyrin amino acid 577 cause autosomal dominant autoinflammatory disease. Ann Rheum Dis 2014;73(2):455‑61. Wang Q, Jin T, Jian S, Han X, Song H, Zhou Q, et al. A dominant pathogenic MEFV mutation causes atypical pyrin-associated periodic syndromes. JCI Insight 2023;e172975.
15. Rowczenio DM, Iancu DS, Trojer H, Gilbertson JA, Gillmore JD, Wechalekar AD, et al. Autosomal dominant familial mediterranean fever in Northern European Caucasians associated with deletion of p.M694 residue-a case series and genetic exploration. Rheumatology (Oxford) 2017;56(2):209‑13.
16. Moghaddas F, Llamas R, De Nardo D, Martinez-Banaclocha H, Martinez-Garcia JJ, Mesa-Del-Castillo P, et al. A novel pyrin-associated autoinflammation with neutrophilic dermatosis mutation further defines 14-3-3 binding of pyrin and distinction to familial mediterranean Fever. Ann Rheum Dis 2017;76(12):2085‑94.
17. Zhang SQ, Jiang T, Li M, Zhang X, Ren YQ, Wei SC, et al. Exome sequencing identifies MVK mutations in disseminated superficial actinic porokeratosis. Nat Genet 2012;44(10):1156‑60.
18. Touitou I. Twists and turns of the genetic story of mevalonate kinase-associated diseases: A review. Genes Dis 2022;9(4):1000‑7.
19. Hoffman HM, Mueller JL, Broide DH, Wanderer AA, Kolodner RD. Mutation of a new gene encoding a putative pyrin-like protein causes familial cold autoinflammatory syndrome and Muckle-Wells syndrome. Nat Genet 2001;29(3):301‑5.
20. McDermott MF, Aksentijevich I, Galon J, McDermott EM, Ogunkolade BW, Centola M, et al. Germline mutations in the extracellular domains of the 55 kDa TNF receptor, TNFR1, define a family of dominantly inherited autoinflammatory syndromes. Cell 1999;97(1):133‑44.
21. Labrousse M, Kevorkian-Verguet C, Boursier G, Rowczenio D, Maurier F, Lazaro E, et al. Mosaicism in autoinflammatory diseases: Cryopyrin-associated periodic syndromes (CAPS) and beyond. A systematic review. Crit Rev Clin Lab Sci 2018;55(6):432‑42.
22. de Jesus AA, Hou Y, Brooks S, Malle L, Biancotto A, Huang Y, et al. Distinct interferon signatures and cytokine patterns define additional systemic autoinflammatory diseases. J Clin Invest 2020;130(4):1669‑82.
23. Lee Y, Wessel AW, Xu J, Reinke JG, Lee E, Kim SM, et al. Genetically programmed alternative splicing of NEMO mediates an autoinflammatory disease phenotype. J Clin Invest 2022;132(6):e128808.
24. Beck DB, Ferrada MA, Sikora KA, Ombrello AK, Collins JC, Pei W, et al. Somatic mutations in UBA1 and severe adult-onset autoinflammatory disease. N Engl J Med 2020;383(27):2628‑38.
25. Agarwal AK, Xing C, DeMartino GN, Mizrachi D, Hernandez MD, Sousa AB, et al. PSMB8 encoding the β5i proteasome subunit is mutated in joint contractures, muscle atrophy, microcytic anemia, and panniculitis-induced lipodystrophy syndrome. Am J Hum Genet 2010;87(6):866‑72.
26. Arima K, Kinoshita A, Mishima H, Kanazawa N, Kaneko T, Mizushima T, et al. Proteasome assembly defect due to a proteasome subunit beta type 8 (PSMB8) mutation causes the autoinflammatory disorder, Nakajo-Nishimura syndrome. Proc Natl Acad Sci USA 2011;108(36):14914‑9.
27. Brehm A, Liu Y, Sheikh A, Marrero B, Omoyinmi E, Zhou Q, et al. Additive loss-of-function proteasome subunit mutations in CANDLE/PRAAS patients promote type I IFN production. J Clin Invest 2015;125(11):4196‑211.
28. Krawiec P, Pawłowska-Kamieniak A, Pac-Kożuchowska E. Interleukin 10 and interleukin 10 receptor in paediatric inflammatory bowel disease: From bench to bedside lesson. J Inflamm (Lond) 2021;18(1):13.
29. Blaydon DC, Biancheri P, Di WL, Plagnol V, Cabral RM, Brooke MA, et al. Inflammatory skin and bowel disease linked to ADAM17 deletion. N Engl J Med 2011;365(16):1502‑8.
30. Zhou Q, Wang H, Schwartz DM, Stoffels M, Park YH, Zhang Y, et al. Loss-of-function mutations in TNFAIP3 leading to A20 haploinsufficiency cause an early-onset autoinflammatory disease. Nat Genet 2016;48(1):67‑73.
31. Koné-Paut I, Sanchez E, Le Quellec A, Manna R, Touitou I. Autoinflammatory gene mutations in Behçet’s disease. Ann Rheum Dis 2007;66(6):832‑4.
32. Wakil SM, Monies DM, Abouelhoda M, Al-Tassan N, Al-Dusery H, Naim EA, et al. Association of a mutation in LACC1 with a monogenic form of systemic juvenile idiopathic arthritis. Arthritis Rheumatol 2015;67(1):288‑95.
33. Shinar Y, Ceccherini I, Rowczenio D, Aksentijevich I, Arostegui J, Ben-Chétrit E, et al. ISSAID/EMQN best practice guidelines for the genetic diagnosis of monogenic autoinflammatory diseases in the next-generation sequencing era. Clin Chem 2020;66(4):525‑36.