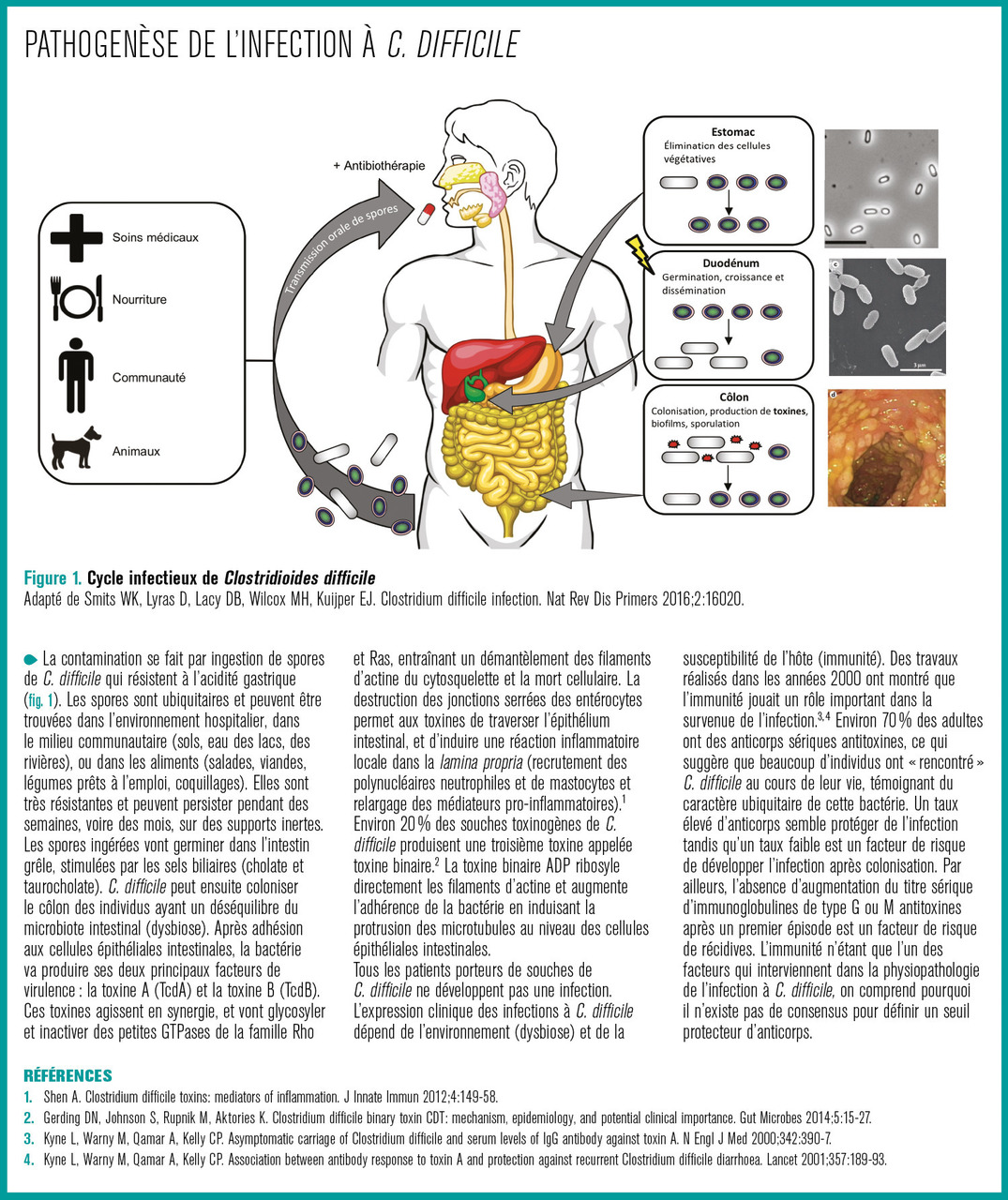
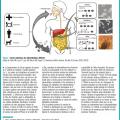
Clostridioides difficile (anciennement appelé Clostridium difficile)1 est une bactérie anaérobie à Gram positif sporulée toxinogène. Depuis la découverte de son rôle dans les colites pseudomembraneuses post-antibiotiques à la fin des années 1970, C. difficile est devenue un entéropathogène majeur, responsable de 15 à 25 % des cas de diarrhées post-antibiotiques et de 95 % des cas de colite pseudomembraneuse.2, 3 Cette bactérie est considérée aujourd’hui non seulement comme le principal agent des diarrhées associées aux soins chez l’adulte4 mais aussi comme un pathogène fréquemment impliqué dans les diarrhées d’origine communautaire.5
L’expression clinique des infections à C. difficile varie de la diarrhée banale sans signes généraux à la colite grave pouvant se compliquer de mégacôlon toxique, de perforation digestive, de choc septique, voire de décès. Ces complications sont rares (< 5 %), et leur prise en charge est d’ordre réanimatoire ou chirurgical (colectomie). Les récidives sont une autre complication, beaucoup plus fréquente, de ces infections : en effet, environ 25 % des patients vont développer une récidive dans les 2 mois qui suivent un épisode initial, et un patient qui a une première récidive augmente son risque de récidives ultérieures.6
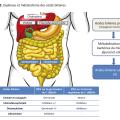
Les facteurs de risque d’infections à C. difficile sont à présent bien connus7, 8 et sont présentés dans letableau ci-contre .
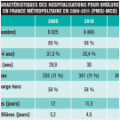
La fréquence des infections à C. difficile n’a cessé d’augmenter au cours de ces 15 dernières années. En Europe, leur incidence moyenne est passée de 4,1 pour 10 000 journées d’hospitalisation en 2008 à 7,9 en 2012-2013.9, 10 Environ 20 à 25 % des infections à C. difficile ne sont pas diagnostiquées en raison soit du manque de sensibilisation des médecins qui ne prescrivent pas spécifiquement la recherche de cette bactérie, soit du défaut de sensibilité des méthodes ou stratégies diagnostiques utilisées.10 En France, les données du réseau Bactéries multirésistantes-Réseau d’alerte d’investigation et de surveillance des infections nosocomiales (BMR-RAISIN) de 2016 ont permis d’estimer l’incidence annuelle des infections à C. difficile à 3,6 pour 10 000 journées d’hospitalisation.11 L’analyse de la base de données Programme de médicalisation des systèmes d’information (PMSI) de 2016 indique qu’environ 20 000 séjours hospitaliers mentionnent une infection à C. difficile comme diagnostic principal ou associé. Des études récentes ont montré que le coût de l’infection à C. difficile en France s’élève à 9 024 € par séjour.12 Par ailleurs, les patients ayant une infection à C. difficile ont une surmortalité à 30 jours et à 1 an significativement supérieure (odds ratio : 1,58 ; p < 0,001) à celle de témoins sans infection à C. difficile appariés sur un score de propension (mêmes comorbidités, même âge, même motif d’hospitalisation).13
L’expression clinique des infections à C. difficile varie de la diarrhée banale sans signes généraux à la colite grave pouvant se compliquer de mégacôlon toxique, de perforation digestive, de choc septique, voire de décès. Ces complications sont rares (< 5 %), et leur prise en charge est d’ordre réanimatoire ou chirurgical (colectomie). Les récidives sont une autre complication, beaucoup plus fréquente, de ces infections : en effet, environ 25 % des patients vont développer une récidive dans les 2 mois qui suivent un épisode initial, et un patient qui a une première récidive augmente son risque de récidives ultérieures.6
Les facteurs de risque d’infections à C. difficile sont à présent bien connus7, 8 et sont présentés dans le
La fréquence des infections à C. difficile n’a cessé d’augmenter au cours de ces 15 dernières années. En Europe, leur incidence moyenne est passée de 4,1 pour 10 000 journées d’hospitalisation en 2008 à 7,9 en 2012-2013.9, 10 Environ 20 à 25 % des infections à C. difficile ne sont pas diagnostiquées en raison soit du manque de sensibilisation des médecins qui ne prescrivent pas spécifiquement la recherche de cette bactérie, soit du défaut de sensibilité des méthodes ou stratégies diagnostiques utilisées.10 En France, les données du réseau Bactéries multirésistantes-Réseau d’alerte d’investigation et de surveillance des infections nosocomiales (BMR-RAISIN) de 2016 ont permis d’estimer l’incidence annuelle des infections à C. difficile à 3,6 pour 10 000 journées d’hospitalisation.11 L’analyse de la base de données Programme de médicalisation des systèmes d’information (PMSI) de 2016 indique qu’environ 20 000 séjours hospitaliers mentionnent une infection à C. difficile comme diagnostic principal ou associé. Des études récentes ont montré que le coût de l’infection à C. difficile en France s’élève à 9 024 € par séjour.12 Par ailleurs, les patients ayant une infection à C. difficile ont une surmortalité à 30 jours et à 1 an significativement supérieure (odds ratio : 1,58 ; p < 0,001) à celle de témoins sans infection à C. difficile appariés sur un score de propension (mêmes comorbidités, même âge, même motif d’hospitalisation).13
Quels liens entre infection à C. difficile et microbiote ?
Résistance à la colonisation
Le tractus gastro-intestinal héberge un des écosystèmes microbiens (ou microbiote) les plus denses et variés,14 composé d’environ 1013 bactéries appartenant à plusieurs centaines d’espèces différentes réparties en deux phyla dominants chez l’adulte (Firmicutes, Bacteroidetes) et plusieurs phyla minoritaires (Actinobacteria, Proteobacteria et Verrucomicrobia). Ce microbiote joue un rôle physiologique pivot dans la maturation du tube digestif, le développement de la réponse immunitaire, et le métabolisme intestinal. Certains genres comme Bacteroides, Bifidobacterium, Clostridium clusters XIVa et IVa, Eubacterium, Faecalibacterium, Lactobacillus et Roseburia ont été associés à un effet bénéfique sur la santé.15 Le microbiote intestinal permet à l’hôte d’être protégé contre les bactéries exogènes pathogènes, ce phénomène étant communément appelé la « résistance à la colonisation ». Cette protection s’exerce en empêchant la bactérie pathogène de s’implanter par un mécanisme de compétition à la fois spatiale et nutritionnelle.
Dans l’infection à C. difficile, le facteur déclenchant est dans plus de 80 % des cas l’antibiothérapie. On comprend alors pourquoi ces infections sont plus fréquentes en milieu hospitalier où près de 15 % des patients sont exposés aux antibiotiques et à un environnement fortement contaminé par les spores. Les antibiotiques vont déséquilibrer le microbiote intestinal et réduire sa richesse et sa diversité, rendant l’hôte sensible à la colonisation puis à l’infection par C. difficile. La dysbiose induite par l’antibiothérapie dépend du spectre de l’antibiotique. Par exemple, l’administration d’amoxicilline-acide clavulanique est associée à une diminution importante des groupes C. coccoides et C. leptum.16 La clindamycine entraîne une diminution persistante du nombre et de la diversité des Bacteroides.17 Après l’arrêt de l’antibiotique, le microbiote retrouve son état initial (résilience) dans un délai qui varie selon les antibiotiques et qui peut aller de quelques semaines à quelques mois.
De nombreuses études ont montré une diminution de la richesse et de la diversité microbienne intestinale chez les patients atteints d’infection à C. difficile ou de récidives par rapport à des patients porteurs asymptomatiques de C. difficile ou des témoins négatifs.18-21 Cette dysbiose est habituellement caractérisée par une diminution des Bacteroides, Bifidobacterium et bactéries productrices de butyrate (Ruminococcaceae, Lachnospiraceae) et une augmentation des γ-Proteobacteria (entérobacteries) et Enterococcus. L’analyse à l’aide d’un modèle de régression logistique du microbiote intestinal de 338 patients a révélé que les selles des patients infectés par C. difficile étaient enrichies en espèces appartenant aux Enterococcaceae, Enterobacteriaceae, Erysipelotrichaceae et Lachnospiraceae et déplétées en certaines espèces appartenant aux Ruminococcaceae, Porphyromonadaceae, Lachnospiraceae et Bacteroides par rapport aux contrôles sains ou aux patients diarrhéiques sans infection à C. difficile.22 D’autres études ont montré une déplétion en certaines espèces bactériennes telle la famille des Clostridiales incertae Sedis XI chez les patients qui développent une infection à C. difficile comparés à des témoins.23 Une équipe24 a comparé le microbiote de 14 enfants âgés de 28 à 48 mois à celui de 43 témoins sains appariés sur l’âge. Comme chez l’adulte, ils ont montré une diversité microbienne significativement inférieure chez les enfants infectés comparé aux témoins. Une augmentation du ratio Firmicutes/Bacteroidetes a été mise en évidence chez les enfants infectés avec une diminution des bactéries productrices de butyrate (Ruminococcaceae incluant Faecalibacterium, Lachnospiraceae et Porphyromonadaceae) et une augmentation des Enterococcaceae, Streptococcaceae, Lactobacillaceae, Actinomycetaceae produisant du lactate.
Pour identifier plus précisément les taxons impliqués dans la résistance à la colonisation par C. difficile, une autre équipe25 a traité des souris par différents antibiotiques pour générer des communautés microbiennes dysbiotiques, puis ont infecté les animaux par des spores de C. difficile. En analysant leur microbiote par séquençage haut débit de l’ARN16S, ils ont ainsi montré que les Escherichia et les Streptococcus étaient associés à un haut niveau de colonisation tandis que les Lachnospiraceae, Porphyromonadaceae, Lactobacillus et Alistipes étaient associés à un faible niveau. Une stratégie comparable26 a permis d’identifier, en comparant à l’aide d’un modèle mathématique le microbiote humain et celui de souris exposées à différents antibiotiques et ayant une sensibilité ou une résistance à l’infection à C. difficile, quatre espèces associées à la résistance à la colonisation : Clostridium scindens, Pseudoflavonifractor capillosus, Blautia hansenii et Barnesiella intestinihominis ; un mélange de ces espèces a ensuite été testé sur un modèle animal d’infection à C. difficile, montrant que C. scindens, seul ou en association, protégeait les animaux de l’infection à C. difficile, suggérant que certaines bactéries spécifiques jouent un rôle prépondérant dans la résistance à la colonisation.
Dans l’infection à C. difficile, le facteur déclenchant est dans plus de 80 % des cas l’antibiothérapie. On comprend alors pourquoi ces infections sont plus fréquentes en milieu hospitalier où près de 15 % des patients sont exposés aux antibiotiques et à un environnement fortement contaminé par les spores. Les antibiotiques vont déséquilibrer le microbiote intestinal et réduire sa richesse et sa diversité, rendant l’hôte sensible à la colonisation puis à l’infection par C. difficile. La dysbiose induite par l’antibiothérapie dépend du spectre de l’antibiotique. Par exemple, l’administration d’amoxicilline-acide clavulanique est associée à une diminution importante des groupes C. coccoides et C. leptum.16 La clindamycine entraîne une diminution persistante du nombre et de la diversité des Bacteroides.17 Après l’arrêt de l’antibiotique, le microbiote retrouve son état initial (résilience) dans un délai qui varie selon les antibiotiques et qui peut aller de quelques semaines à quelques mois.
De nombreuses études ont montré une diminution de la richesse et de la diversité microbienne intestinale chez les patients atteints d’infection à C. difficile ou de récidives par rapport à des patients porteurs asymptomatiques de C. difficile ou des témoins négatifs.18-21 Cette dysbiose est habituellement caractérisée par une diminution des Bacteroides, Bifidobacterium et bactéries productrices de butyrate (Ruminococcaceae, Lachnospiraceae) et une augmentation des γ-Proteobacteria (entérobacteries) et Enterococcus. L’analyse à l’aide d’un modèle de régression logistique du microbiote intestinal de 338 patients a révélé que les selles des patients infectés par C. difficile étaient enrichies en espèces appartenant aux Enterococcaceae, Enterobacteriaceae, Erysipelotrichaceae et Lachnospiraceae et déplétées en certaines espèces appartenant aux Ruminococcaceae, Porphyromonadaceae, Lachnospiraceae et Bacteroides par rapport aux contrôles sains ou aux patients diarrhéiques sans infection à C. difficile.22 D’autres études ont montré une déplétion en certaines espèces bactériennes telle la famille des Clostridiales incertae Sedis XI chez les patients qui développent une infection à C. difficile comparés à des témoins.23 Une équipe24 a comparé le microbiote de 14 enfants âgés de 28 à 48 mois à celui de 43 témoins sains appariés sur l’âge. Comme chez l’adulte, ils ont montré une diversité microbienne significativement inférieure chez les enfants infectés comparé aux témoins. Une augmentation du ratio Firmicutes/Bacteroidetes a été mise en évidence chez les enfants infectés avec une diminution des bactéries productrices de butyrate (Ruminococcaceae incluant Faecalibacterium, Lachnospiraceae et Porphyromonadaceae) et une augmentation des Enterococcaceae, Streptococcaceae, Lactobacillaceae, Actinomycetaceae produisant du lactate.
Pour identifier plus précisément les taxons impliqués dans la résistance à la colonisation par C. difficile, une autre équipe25 a traité des souris par différents antibiotiques pour générer des communautés microbiennes dysbiotiques, puis ont infecté les animaux par des spores de C. difficile. En analysant leur microbiote par séquençage haut débit de l’ARN16S, ils ont ainsi montré que les Escherichia et les Streptococcus étaient associés à un haut niveau de colonisation tandis que les Lachnospiraceae, Porphyromonadaceae, Lactobacillus et Alistipes étaient associés à un faible niveau. Une stratégie comparable26 a permis d’identifier, en comparant à l’aide d’un modèle mathématique le microbiote humain et celui de souris exposées à différents antibiotiques et ayant une sensibilité ou une résistance à l’infection à C. difficile, quatre espèces associées à la résistance à la colonisation : Clostridium scindens, Pseudoflavonifractor capillosus, Blautia hansenii et Barnesiella intestinihominis ; un mélange de ces espèces a ensuite été testé sur un modèle animal d’infection à C. difficile, montrant que C. scindens, seul ou en association, protégeait les animaux de l’infection à C. difficile, suggérant que certaines bactéries spécifiques jouent un rôle prépondérant dans la résistance à la colonisation.
Microbiote, métabolome, métagénome
Les études concernant les structures de populations bactériennes permettent de dégager des tendances sur les classes de micro-organismes associées à l’infection ou à la résistance à l’infection. Les études de métagénomique ou de métabolomique ont apporté un éclairage méca-nistique sur la notion d’effet de barrière.
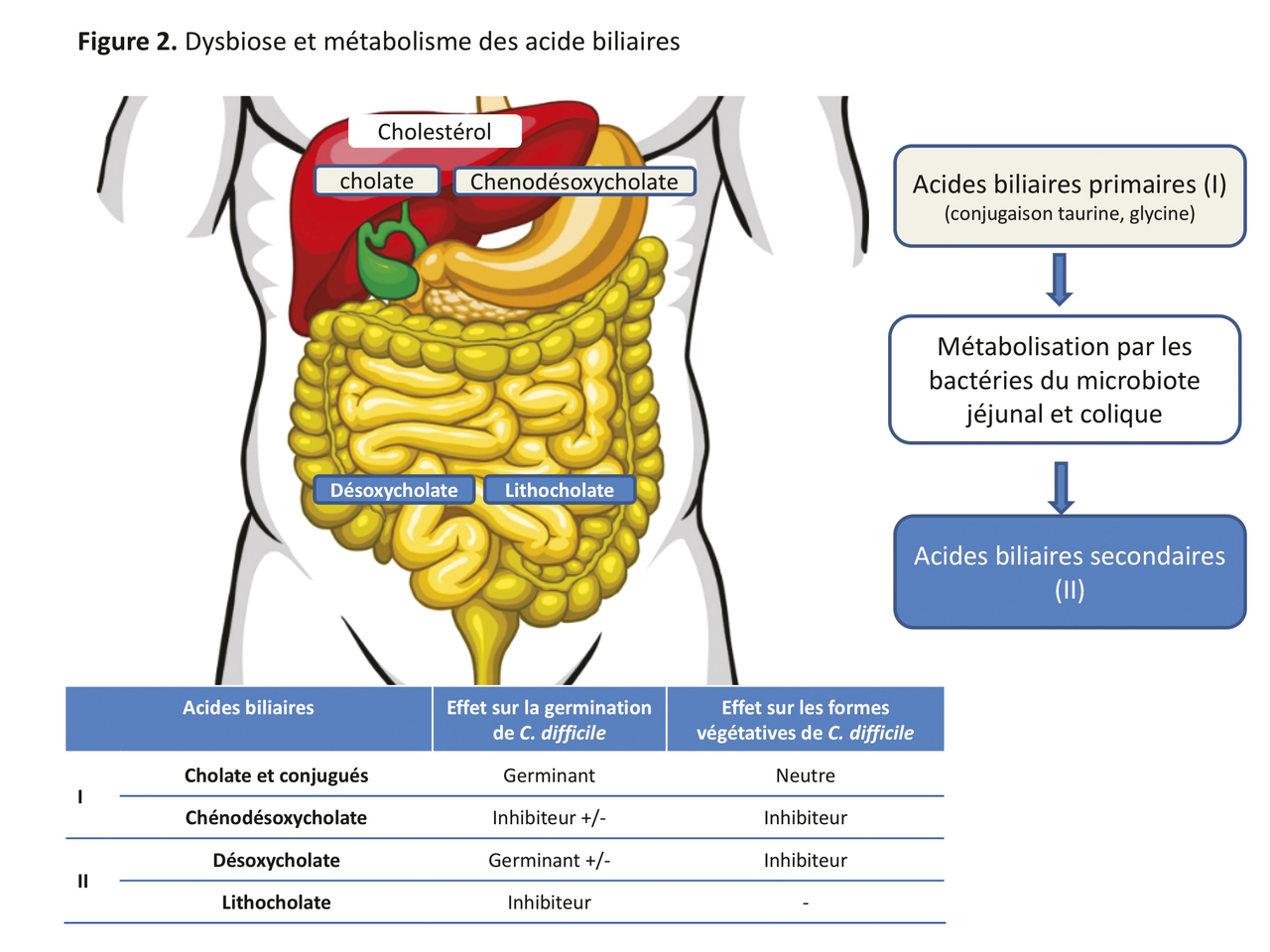
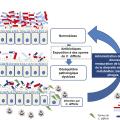
Un rôle important du microbiote intestinal est de pouvoir réguler les acides biliaires primaires et secondaires afin d’inhiber la croissance de C. difficile. On sait depuis des décennies que le taurocholate est un composé qui permet d’induire la germination des spores ; il est habituellement utilisé dans les milieux de culture sélectifs afin de favoriser l’isolement de C. difficile.27 Plus tard, il a été montré que les dérivés de l’acide cholique conjugués à la glycine ou à la taurine activent la germination tandis que les dérivés des acides biliaires secondaires comme le désoxycholate inhibent la germination.28-30 Un traitement antibiotique peut favoriser la croissance de C. difficile en modifiant le métabolisme des acides biliaires (augmentation des acides biliaires germinants et inhibition des acides biliaires inhibant les formes végétatives) [fig. 2 ]. Le transfert des bactéries du contenu cæcal de souris traitées par antibiotiques permet la croissance in vivo de C. difficile, alors que le transfert de contenu cæcal d’animaux non traités augmente la quantité d’acides biliaires secondaires et la résistance à la colonisation.26 L’élévation des acides biliaires secondaires a pu être reproduite chez la souris en lui administrant un cocktail de quatre bactéries, dont Clostridium scindens, qui semble jouer un rôle prépondérant du fait de son enzyme 7-alpha-déshydroxylase qui transforme les acides biliaires primaires en acides biliaires secondaires ;26, 30, 31 cette observation confirme l’hypothèse que certains constituants spécifiques du microbiote pourraient protéger contre l’infection à C. difficile.
D’autres travaux ont montré que les altérations du microbiote induites par les antibiotiques entraînaient des pics d’acide succinique et d’acide sialique qui facilitent la croissance de C. difficile dans l’intestin.32, 33
Un rôle important du microbiote intestinal est de pouvoir réguler les acides biliaires primaires et secondaires afin d’inhiber la croissance de C. difficile. On sait depuis des décennies que le taurocholate est un composé qui permet d’induire la germination des spores ; il est habituellement utilisé dans les milieux de culture sélectifs afin de favoriser l’isolement de C. difficile.27 Plus tard, il a été montré que les dérivés de l’acide cholique conjugués à la glycine ou à la taurine activent la germination tandis que les dérivés des acides biliaires secondaires comme le désoxycholate inhibent la germination.28-30 Un traitement antibiotique peut favoriser la croissance de C. difficile en modifiant le métabolisme des acides biliaires (augmentation des acides biliaires germinants et inhibition des acides biliaires inhibant les formes végétatives) [
D’autres travaux ont montré que les altérations du microbiote induites par les antibiotiques entraînaient des pics d’acide succinique et d’acide sialique qui facilitent la croissance de C. difficile dans l’intestin.32, 33
Quels développements thérapeutiques ?
Transplantation de microbiote fécal et récidives multiples
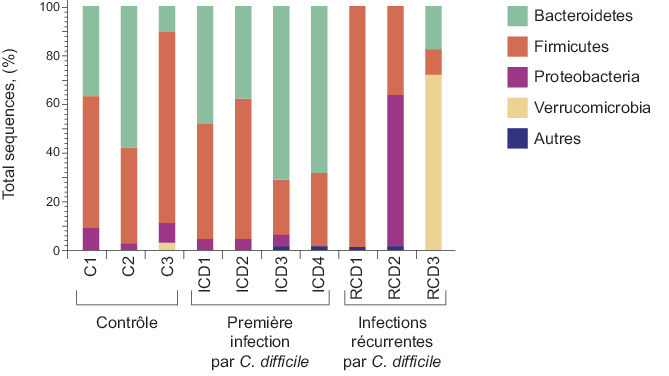
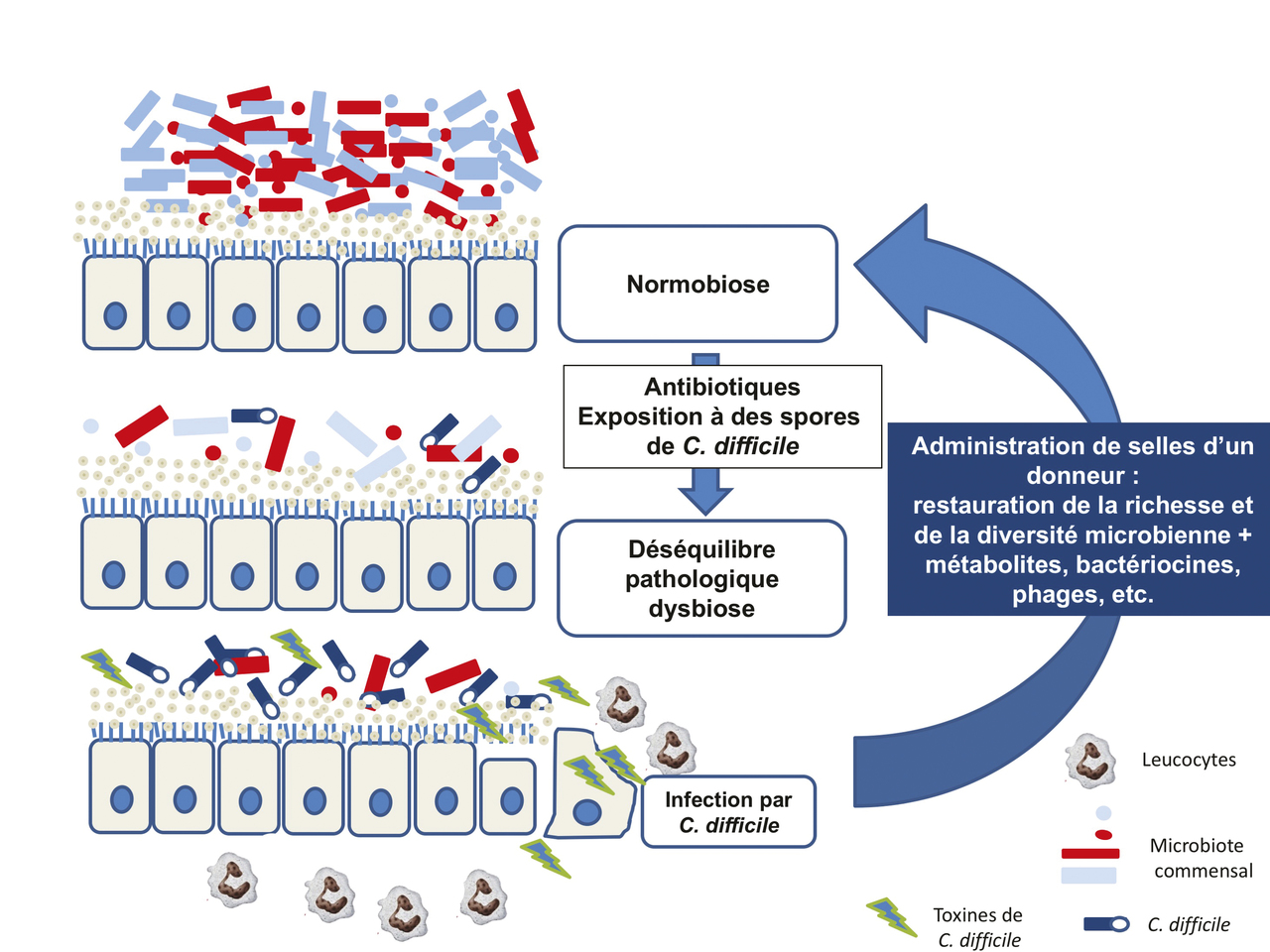
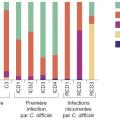
Les récidives multiples sont une complication fréquente des infections à C. difficile et un véritable défi thérapeutique. Elles sont dues à plusieurs facteurs : inaptitude de l’hôte à reconstituer son microbiote intestinal, persistance des spores de C. difficile, ou réponse immunitaire antitoxine suboptimale. Dans une étude déjà ancienne,18 les auteurs avaient montré une diminution très importante de la diversité bactérienne du microbiote fécal chez les patients ayant des récidives multiples comparés à des sujets contrôles indemnes d’infection à C. difficile. En particulier le ratio des deux phyla majoritaires (Bacteroidetes et Firmicutes) était profondément modifié (fig. 3 ). Pendant longtemps, le traitement des récidives a reposé sur l’utilisation d’antibiotiques, qui entretenaient paradoxalement la dysbiose et empêchaient l’hôte de reconsti-tuer son microbiote. La transplantation de microbiote fécal chez les patients souffrant d’infection à C. difficile vise à reconstituer cette diversité microbienne (fig. 4 ). Cette stratégie thérapeutique connaît un succès croissant depuis la première description en 1958 de son utilisation couronnée de succès chez quatre patients souffrant de colites pseudomembraneuses.34 Le premier essai clinique randomisé sur la transplantation de microbiote fécal dans le traitement des infections à C. difficile récidivantes a été conduit entre 2008 et 2010.35 Trois stratégies thérapeutiques ont été comparées : traitement standard par vancomycine (500 mg per os 4 fois par jour pendant 14 jours), traitement par vancomycine associé à un lavement, ou traitement initial par vancomycine suivi par un lavement et une transplantation de microbiote par des selles fraîches administrées par sonde nasoduodénale. Cet essai a montré une supériorité significative de la transplantation en comparaison aux deux autres bras de l’étude (94 % de guérison vs 31 % et 23 % pour respectivement les bras vancomycine seule et vancomycine + lavement). De plus, l’indice de Simpson estimant la diversité du microbiote fécal, initialement bas chez les patients en récidives multiples, était significativement augmenté après transplantation (57 vs 179 ; p < 0,001). Les changements quantitatifs observés incluaient une augmentation des espèces de Bacteroidetes et Clostridium clusters IV et XIVa, ainsi qu’une diminution des Proteobacteria. Plusieurs études ont ensuite montré que la normalisation de la composition du microbiote du receveur était très rapide, que sa composition était très similaire à celle du donneur à court terme après la transplantation fécale et restait similaire à celle d’autres sujets sains jusqu’au moins 151 jours après la transplantation.36, 37 D’autres essais cliniques sont venus ensuite conforter ces résultats.38, 39 Dans une méta-analyse récente comprenant sept essais randomisés contrôlés et 30 séries de cas, la transplantation de microbiote fécal était plus efficace que la vancomycine pour les infections récidivantes à C. difficile (risque relatif : 0,23 ; intervalle de confiance [IC] à 95 % : 0,07-0,80) et le taux de succès clinique était de 92 % (IC à 95 % : 89-94).40
Une observation récente41 a amené un éclairage nouveau sur le mécanisme d’action de la transplantation de microbiote fécal. Les auteurs ont traité cinq patients souffrant d’infection à C. difficile récidivante par un filtrat stérile de selles de donneurs sains. Les symptômes des cinq patients se sont très rapidement améliorés, et aucune récidive n’a été rapportée dans les 6 mois qui ont suivi. Si ces résultats sont confirmés dans une étude randomisée, cela pourrait suggérer que l’efficacité de la transplantation de microbiote fécal pourrait ne pas être liée directement aux bactéries viables mais à la présence de composés dans les selles tels des protéines, des bactériocines, des bactériophages, des produits du métabolisme ou des oligonucléotides/ADN.
Une observation récente41 a amené un éclairage nouveau sur le mécanisme d’action de la transplantation de microbiote fécal. Les auteurs ont traité cinq patients souffrant d’infection à C. difficile récidivante par un filtrat stérile de selles de donneurs sains. Les symptômes des cinq patients se sont très rapidement améliorés, et aucune récidive n’a été rapportée dans les 6 mois qui ont suivi. Si ces résultats sont confirmés dans une étude randomisée, cela pourrait suggérer que l’efficacité de la transplantation de microbiote fécal pourrait ne pas être liée directement aux bactéries viables mais à la présence de composés dans les selles tels des protéines, des bactériocines, des bactériophages, des produits du métabolisme ou des oligonucléotides/ADN.
Autres approches
D’autres approches thérapeutiques ont été proposées, fondées sur le concept de l’administration de bactéries ou d’un cocktail de bactéries qui pourraient restaurer la résistance à la colonisation, tout en garantissant une meilleure sécurité et une plus grande standardisation par rapport à l’administration de selles.
Les pionniers dans ce domaine ont administré dès 1989 un mélange de 10 souches bactériennes isolées de selles de donneurs sains à cinq patients atteints de récidives d’infection à C. difficile. Les patients ont rapidement eu une amélioration clinique, une éradication de C. difficile et de leurs toxines et une repopulation des selles par des Bacteroides sp. Les auteurs suggèrent que l’effet bénéfique est lié à la capacité de trois souches (Peptostreptococcus productus, Clostridium bifermentans et E. coli) à inhiber in vitro la croissance de C. difficile.42 Plus récemment, le RBX2660 (Rebiotix), composé d’une suspension de bactéries administrées par lavement, a fait l’objet d’un essai de phase II chez 31 patients souffrant de récidives multiples d’infection à C. difficile. Le taux de guérison à 6 mois était de 61 % contre 45 % pour le groupe contrôle.43
Une autre approche consiste à administrer des selles de donneurs traitées par alcool afin de ne sélectionner que des bactéries sporulées (SER-109, Seres). Un essai clinique ouvert de phase I incluant 30 patients ayant des récidives d’infection à C. difficile a montré une greffe très rapide des souches du donneur et une guérison clinique du receveur de 86,7 %.44 Ces résultats positifs n’ont cependant pas été confirmés lors d’un essai randomisé double aveugle contre placebo de phase II, et le développement du produit a été interrompu.
Deux patients en échec de traitement de récidives ont été traités avec succès par un mélange de 33 souches bactériennes purifiées provenant de donneurs sains et administrées par voie endoscopique.45 L’analyse des selles des receveurs par séquençage haut débit de l’ARN 16S a montré que les souches administrées étaient rares avant leur administration mais représentaient jusqu’à 25 % des séquences 6 mois après l’intervention.
Un approche originale repose sur l’administration de souches non toxinogènes (donc non pathogènes) de C. difficiles, avec une action bénéfique sur le système immunitaire, et une action inhibitrice de la croissance de souches toxinogènes responsables d’infections. Un essai de phase II randomisés en double aveugle contre placebo a été réalisé auprès de 173 patients avec un premier épisode ou une récidive d’infection à C. difficile traitée par vancomycine ou métronidazole et recevant 104 à 107 spores pendant 7-14 jours. Le taux de colonisation variait de 63 à 71 %. Les résultats ont montré que le taux de récidives était significativement plus faible chez les patients colonisés par les souches non toxinogènes comparés aux patients non colonisés (2 % vs 31 %; p < 0,001).46
Les pionniers dans ce domaine ont administré dès 1989 un mélange de 10 souches bactériennes isolées de selles de donneurs sains à cinq patients atteints de récidives d’infection à C. difficile. Les patients ont rapidement eu une amélioration clinique, une éradication de C. difficile et de leurs toxines et une repopulation des selles par des Bacteroides sp. Les auteurs suggèrent que l’effet bénéfique est lié à la capacité de trois souches (Peptostreptococcus productus, Clostridium bifermentans et E. coli) à inhiber in vitro la croissance de C. difficile.42 Plus récemment, le RBX2660 (Rebiotix), composé d’une suspension de bactéries administrées par lavement, a fait l’objet d’un essai de phase II chez 31 patients souffrant de récidives multiples d’infection à C. difficile. Le taux de guérison à 6 mois était de 61 % contre 45 % pour le groupe contrôle.43
Une autre approche consiste à administrer des selles de donneurs traitées par alcool afin de ne sélectionner que des bactéries sporulées (SER-109, Seres). Un essai clinique ouvert de phase I incluant 30 patients ayant des récidives d’infection à C. difficile a montré une greffe très rapide des souches du donneur et une guérison clinique du receveur de 86,7 %.44 Ces résultats positifs n’ont cependant pas été confirmés lors d’un essai randomisé double aveugle contre placebo de phase II, et le développement du produit a été interrompu.
Deux patients en échec de traitement de récidives ont été traités avec succès par un mélange de 33 souches bactériennes purifiées provenant de donneurs sains et administrées par voie endoscopique.45 L’analyse des selles des receveurs par séquençage haut débit de l’ARN 16S a montré que les souches administrées étaient rares avant leur administration mais représentaient jusqu’à 25 % des séquences 6 mois après l’intervention.
Un approche originale repose sur l’administration de souches non toxinogènes (donc non pathogènes) de C. difficiles, avec une action bénéfique sur le système immunitaire, et une action inhibitrice de la croissance de souches toxinogènes responsables d’infections. Un essai de phase II randomisés en double aveugle contre placebo a été réalisé auprès de 173 patients avec un premier épisode ou une récidive d’infection à C. difficile traitée par vancomycine ou métronidazole et recevant 104 à 107 spores pendant 7-14 jours. Le taux de colonisation variait de 63 à 71 %. Les résultats ont montré que le taux de récidives était significativement plus faible chez les patients colonisés par les souches non toxinogènes comparés aux patients non colonisés (2 % vs 31 %; p < 0,001).46
Bientôt d’autres biothérapies…
Les avancées technologiques (séquençage haut débit ARN 16S, métagénomique, métabolomique) ont permis des progrès notables dans la connaissance de la structure des communautés bactériennes intestinales et la compréhension de la résistance à la colonisation par C. difficile. La restauration de la diversité microbienne par transplantation de microbiote fécal a connu ces dernières années un essor considérable dans le traitement des récidives multiples d’infection à C. difficile. D’autres approches de « biothérapies », moins à risque que la transplantation fécale, sont à l’étude.
Références
1. Lawson PA, Citron DM, Tyrrell KL, Finegold SM. Reclassification of Clostridium difficile as Clostridioides difficile (Hall and O'Toole 1935) Prevot 1938. Anaerobe 2016;40:95-9.
2. Rupnik M, Wilcox MH, Gerding DN. Clostridium difficile infection: new developments in epidemiology and pathogenesis. Nat Rev Microbiol 2009;7:526-36.
3. Barbut F, Jones G, Eckert C. Epidemiology and control of Clostridium difficile infections in healthcare settings: an update. Curr Opin Infect Dis 2011;24:370-6.
4. Magill SS, Edwards JR, Fridkin SK. Emerging Infections Program Healthcare-Associated I, Antimicrobial Use Prevalence Survey T. Survey of health care-associated infections. N Engl J Med 2014;370:2542-3.
5. Barbut F, Day N, Bouee S, et al. Toxigenic Clostridium difficile carriage in general practice: results of a laboratory-based cohort study. Clin Microbiol Infect 2019;25:588-94.
6. Kelly CP. Can we identify patients at high risk of recurrent Clostridium difficile infection? Clin Microbiol Infec 2012;18(Suppl 6):21-7.
7. McFarland LV, Mulligan ME, Kwok RY, Stamm WE. Nosocomial acquisition of Clostridium difficile infection. N Engl J Med 1989;320:204-10.
8. Loo VG, Bourgault AM, Poirier L, et al. Host and pathogen factors for Clostridium difficile infection and colonization. N Engl J Med 2011;365:1693-703.
9. Bauer MP, Notermans DW, van Benthem BH, et al. Clostridium difficile infection in Europe: a hospital-based survey. Lancet 2011;377:63-73.
10. Davies KA, Longshaw CM, Davis GL, et al. Underdiagnosis of Clostridium difficile across Europe: the European, multicentre, prospective, biannual, point-prevalence study of Clostridium difficile infection in hospitalised patients with diarrhoea (EUCLID). Lancet Infect Dis 2014;14:1208-19.
11. Cotinat et al., Euro-surveillance, 2019, sous presse.
12. Le Monnier A, Duburcq A, Zahar JR, et al. Hospital cost of Clostridium difficile infection including the contribution of recurrences in French acute-care hospitals. J Hosp Infect 2015;91:117-22.
13. Barbut F, Bouee S, Longepierre L, Goldberg M, Bensoussan C, Levy-Bachelot L. Excess mortality between 2007 and 2014 among patients with Clostridium difficile infection: a French health insurance database analysis. J Hosp Infect 2018;98:21-8.
14. Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature 2010;464:59-65.
15. Hollister EB, Gao C, Versalovic J. Compositional and functional features of the gastrointestinal microbiome and their effects on human health. Gastroenterology 2014;146:1449-58.
16. Young VB, Schmidt TM. Antibiotic-associated diarrhea accompanied by large-scale alterations in the composition of the fecal microbiota. J Clin Microbiol 2004;42:1203-6.
17. Jernberg C, Lofmark S, Edlund C, Jansson JK. Long-term impacts of antibiotic exposure on the human intestinal microbiota. Microbiology 2010;156(Pt 11):3216-23.
18. Chang JY, Antonopoulos DA, Kalra A, et al. Decreased diversity of the fecal Microbiome in recurrent Clostridium difficile-associated diarrhea. J Infect Dis 2008;197:435-8.
19. Rousseau C, Poilane I, De Pontual L, Maherault AC, Le Monnier A, Collignon A. Clostridium difficile carriage in healthy infants in the community: a potential reservoir for pathogenic strains. Clin Infect Dis 2012;55:1209-15.
20. Antharam VC, Li EC, Ishmael A, et al. Intestinal dysbiosis and depletion of butyrogenic bacteria in Clostridium difficile infection and nosocomial diarrhea. J Clin Microbiol 2013;51:2884-92.
21. Rea MC, O'Sullivan O, Shanahan F, et al. Clostridium difficile carriage in elderly subjects and associated changes in the intestinal microbiota. J Clin Microbiol 2012;50:867-75.
22. Schubert AM, Rogers MA, Ring C, et al. Microbiome data distinguish patients with Clostridium difficile infection and non-C. difficile-associated diarrhea from healthy controls. MBio. 2014;5(3):e01021-14.
23. Vincent C, Stephens DA, Loo VG, et al. Reductions in intestinal Clostridiales precede the development of nosocomial Clostridium difficile infection. Microbiome 2013;1:18.
24. Ling Z, Liu X, Jia X, et al. Impacts of infection with different toxigenic Clostridium difficile strains on faecal microbiota in children. Sci Rep 2014;4:7485.
25. Schubert AM, Sinani H, Schloss PD. Antibiotic-induced alterations of the murine gut microbiota and subsequent effects on colonization resistance against Clostridium difficile. MBio 2015;6:e00974.
26. Buffie CG, Bucci V, Stein RR, et al. Precision microbiome reconstitution restores bile acid mediated resistance to Clostridium difficile. Nature 2015;517:205-8.
27. Wilson KH, Kennedy MJ, Fekety FR. Use of sodium taurocholate to enhance spore recovery on a medium selective for Clostridium difficile. J Clin Microbiol 1982;15:443-6.
28. Savidge T, Sorg JA. Role of bile in infectious disease: the gall of 7alpha-dehydroxylating gut bacteria. Cell Chem Biol 2019;26:1-3.
29. Sorg JA, Sonenshein AL. Bile salts and glycine as cogerminants for Clostridium difficile spores. J Bacteriol 2008;190:2505-12.
30. Sorg JA, Sonenshein AL. Inhibiting the initiation of Clostridium difficile spore germination using analogs of chenodeoxycholic acid, a bile acid. J Bacteriol 2010;192:4983-90.
31. Amrane S, Raoult D, Lagier JC. Metagenomics, culturomics, and the human gut microbiota. Expert Rev Anti Infect Ther 2018;16:373-5.
32. Ferreyra JA, Wu KJ, Hryckowian AJ, Bouley DM, Weimer BC, Sonnenburg JL. Gut microbiota-produced succinate promotes C. difficile infection after antibiotic treatment or motility disturbance. Cell Host Microbe 2014;16:770-7.
33. Ng KM, Ferreyra JA, Higginbottom SK, et al. Microbiota-liberated host sugars facilitate post-antibiotic expansion of enteric pathogens. Nature 2013;502:96-9.
34. Eiseman B, Silen W, Bascom GS, Kauvar AJ. Fecal enema as an adjunct in the treatment of pseudomembranous enterocolitis. Surgery 1958;44:854-9.
35. van Nood E, Vrieze A, Nieuwdorp M, et al. Duodenal infusion of donor feces for recurrent Clostridium difficile. N Engl J Med 2013;368:407-15.
36. Weingarden A, Gonzalez A, Vazquez-Baeza Y, et al. Dynamic changes in short- and long-term bacterial composition following fecal microbiota transplantation for recurrent Clostridium difficile infection. Microbiome 2015;3:10.
37. Shankar V, Hamilton MJ, Khoruts A, et al. Species and genus level resolution analysis of gut microbiota in Clostridium difficile patients following fecal microbiota transplantation. Microbiome 2014;2:13.
38. Cammarota G, Masucci L, Ianiro G, et al. Randomised clinical trial: faecal microbiota transplantation by colonoscopy vs. vancomycin for the treatment of recurrent Clostridium difficile infection. Aliment Pharmacol Ther 2015;41:835-43.
39. Kelly CR, Khoruts A, Staley C, et al. Effect of fecal microbiota transplantation on recurrence in multiply recurrent Clostridium difficile infection: a randomized trial. Ann Intern Med 2016;165:609-16.
40. Quraishi MN, Widlak M, Bhala N, et al. Systematic review with meta-analysis: the efficacy of faecal microbiota transplantation for the treatment of recurrent and refractory Clostridium difficile infection. Aliment Pharmacol Ther 2017;46:479-93.
41. Ott SJ, Waetzig GH, Rehman A, et al. Efficacy of sterile fecal filtrate transfer for treating patients with Clostridium difficile infection. Gastroenterology 2017;152:799-811.
42. Tvede M, Rask-Madsen J. Bacteriotherapy for chronic relapsing Clostridium difficile diarrhoea in six patients. Lancet 1989;1:1156-60.
43. Dubberke ER, Lee CH, Orenstein R, Khanna S, Hecht G, Gerding DN. Results from a randomized, placebo-controlled clinical trial of a RBX2660-A microbiota-based drug for the prevention of recurrent Clostridium difficile infection. Clin Infect Dis 2018;67:1198-204.
44. Khanna S, Pardi DS, Kelly CR, et al. A novel microbiome therapeutic increases gut microbial diversity and prevents recurrent Clostridium difficile infection. J Infect Dis 2016;214:173-81.
45. Petrof EO, Gloor GB, Vanner SJ, et al. Stool substitute transplant therapy for the eradication of Clostridium difficile infection: 'RePOOPulating' the gut. Microbiome 2013;1:3.
46. Gerding DN, Meyer T, Lee C, et al. Administration of spores of nontoxigenic Clostridium difficile strain M3 for prevention of recurrent C. difficile infection: a randomized clinical trial. JAMA 2015;313:1719-27.
2. Rupnik M, Wilcox MH, Gerding DN. Clostridium difficile infection: new developments in epidemiology and pathogenesis. Nat Rev Microbiol 2009;7:526-36.
3. Barbut F, Jones G, Eckert C. Epidemiology and control of Clostridium difficile infections in healthcare settings: an update. Curr Opin Infect Dis 2011;24:370-6.
4. Magill SS, Edwards JR, Fridkin SK. Emerging Infections Program Healthcare-Associated I, Antimicrobial Use Prevalence Survey T. Survey of health care-associated infections. N Engl J Med 2014;370:2542-3.
5. Barbut F, Day N, Bouee S, et al. Toxigenic Clostridium difficile carriage in general practice: results of a laboratory-based cohort study. Clin Microbiol Infect 2019;25:588-94.
6. Kelly CP. Can we identify patients at high risk of recurrent Clostridium difficile infection? Clin Microbiol Infec 2012;18(Suppl 6):21-7.
7. McFarland LV, Mulligan ME, Kwok RY, Stamm WE. Nosocomial acquisition of Clostridium difficile infection. N Engl J Med 1989;320:204-10.
8. Loo VG, Bourgault AM, Poirier L, et al. Host and pathogen factors for Clostridium difficile infection and colonization. N Engl J Med 2011;365:1693-703.
9. Bauer MP, Notermans DW, van Benthem BH, et al. Clostridium difficile infection in Europe: a hospital-based survey. Lancet 2011;377:63-73.
10. Davies KA, Longshaw CM, Davis GL, et al. Underdiagnosis of Clostridium difficile across Europe: the European, multicentre, prospective, biannual, point-prevalence study of Clostridium difficile infection in hospitalised patients with diarrhoea (EUCLID). Lancet Infect Dis 2014;14:1208-19.
11. Cotinat et al., Euro-surveillance, 2019, sous presse.
12. Le Monnier A, Duburcq A, Zahar JR, et al. Hospital cost of Clostridium difficile infection including the contribution of recurrences in French acute-care hospitals. J Hosp Infect 2015;91:117-22.
13. Barbut F, Bouee S, Longepierre L, Goldberg M, Bensoussan C, Levy-Bachelot L. Excess mortality between 2007 and 2014 among patients with Clostridium difficile infection: a French health insurance database analysis. J Hosp Infect 2018;98:21-8.
14. Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature 2010;464:59-65.
15. Hollister EB, Gao C, Versalovic J. Compositional and functional features of the gastrointestinal microbiome and their effects on human health. Gastroenterology 2014;146:1449-58.
16. Young VB, Schmidt TM. Antibiotic-associated diarrhea accompanied by large-scale alterations in the composition of the fecal microbiota. J Clin Microbiol 2004;42:1203-6.
17. Jernberg C, Lofmark S, Edlund C, Jansson JK. Long-term impacts of antibiotic exposure on the human intestinal microbiota. Microbiology 2010;156(Pt 11):3216-23.
18. Chang JY, Antonopoulos DA, Kalra A, et al. Decreased diversity of the fecal Microbiome in recurrent Clostridium difficile-associated diarrhea. J Infect Dis 2008;197:435-8.
19. Rousseau C, Poilane I, De Pontual L, Maherault AC, Le Monnier A, Collignon A. Clostridium difficile carriage in healthy infants in the community: a potential reservoir for pathogenic strains. Clin Infect Dis 2012;55:1209-15.
20. Antharam VC, Li EC, Ishmael A, et al. Intestinal dysbiosis and depletion of butyrogenic bacteria in Clostridium difficile infection and nosocomial diarrhea. J Clin Microbiol 2013;51:2884-92.
21. Rea MC, O'Sullivan O, Shanahan F, et al. Clostridium difficile carriage in elderly subjects and associated changes in the intestinal microbiota. J Clin Microbiol 2012;50:867-75.
22. Schubert AM, Rogers MA, Ring C, et al. Microbiome data distinguish patients with Clostridium difficile infection and non-C. difficile-associated diarrhea from healthy controls. MBio. 2014;5(3):e01021-14.
23. Vincent C, Stephens DA, Loo VG, et al. Reductions in intestinal Clostridiales precede the development of nosocomial Clostridium difficile infection. Microbiome 2013;1:18.
24. Ling Z, Liu X, Jia X, et al. Impacts of infection with different toxigenic Clostridium difficile strains on faecal microbiota in children. Sci Rep 2014;4:7485.
25. Schubert AM, Sinani H, Schloss PD. Antibiotic-induced alterations of the murine gut microbiota and subsequent effects on colonization resistance against Clostridium difficile. MBio 2015;6:e00974.
26. Buffie CG, Bucci V, Stein RR, et al. Precision microbiome reconstitution restores bile acid mediated resistance to Clostridium difficile. Nature 2015;517:205-8.
27. Wilson KH, Kennedy MJ, Fekety FR. Use of sodium taurocholate to enhance spore recovery on a medium selective for Clostridium difficile. J Clin Microbiol 1982;15:443-6.
28. Savidge T, Sorg JA. Role of bile in infectious disease: the gall of 7alpha-dehydroxylating gut bacteria. Cell Chem Biol 2019;26:1-3.
29. Sorg JA, Sonenshein AL. Bile salts and glycine as cogerminants for Clostridium difficile spores. J Bacteriol 2008;190:2505-12.
30. Sorg JA, Sonenshein AL. Inhibiting the initiation of Clostridium difficile spore germination using analogs of chenodeoxycholic acid, a bile acid. J Bacteriol 2010;192:4983-90.
31. Amrane S, Raoult D, Lagier JC. Metagenomics, culturomics, and the human gut microbiota. Expert Rev Anti Infect Ther 2018;16:373-5.
32. Ferreyra JA, Wu KJ, Hryckowian AJ, Bouley DM, Weimer BC, Sonnenburg JL. Gut microbiota-produced succinate promotes C. difficile infection after antibiotic treatment or motility disturbance. Cell Host Microbe 2014;16:770-7.
33. Ng KM, Ferreyra JA, Higginbottom SK, et al. Microbiota-liberated host sugars facilitate post-antibiotic expansion of enteric pathogens. Nature 2013;502:96-9.
34. Eiseman B, Silen W, Bascom GS, Kauvar AJ. Fecal enema as an adjunct in the treatment of pseudomembranous enterocolitis. Surgery 1958;44:854-9.
35. van Nood E, Vrieze A, Nieuwdorp M, et al. Duodenal infusion of donor feces for recurrent Clostridium difficile. N Engl J Med 2013;368:407-15.
36. Weingarden A, Gonzalez A, Vazquez-Baeza Y, et al. Dynamic changes in short- and long-term bacterial composition following fecal microbiota transplantation for recurrent Clostridium difficile infection. Microbiome 2015;3:10.
37. Shankar V, Hamilton MJ, Khoruts A, et al. Species and genus level resolution analysis of gut microbiota in Clostridium difficile patients following fecal microbiota transplantation. Microbiome 2014;2:13.
38. Cammarota G, Masucci L, Ianiro G, et al. Randomised clinical trial: faecal microbiota transplantation by colonoscopy vs. vancomycin for the treatment of recurrent Clostridium difficile infection. Aliment Pharmacol Ther 2015;41:835-43.
39. Kelly CR, Khoruts A, Staley C, et al. Effect of fecal microbiota transplantation on recurrence in multiply recurrent Clostridium difficile infection: a randomized trial. Ann Intern Med 2016;165:609-16.
40. Quraishi MN, Widlak M, Bhala N, et al. Systematic review with meta-analysis: the efficacy of faecal microbiota transplantation for the treatment of recurrent and refractory Clostridium difficile infection. Aliment Pharmacol Ther 2017;46:479-93.
41. Ott SJ, Waetzig GH, Rehman A, et al. Efficacy of sterile fecal filtrate transfer for treating patients with Clostridium difficile infection. Gastroenterology 2017;152:799-811.
42. Tvede M, Rask-Madsen J. Bacteriotherapy for chronic relapsing Clostridium difficile diarrhoea in six patients. Lancet 1989;1:1156-60.
43. Dubberke ER, Lee CH, Orenstein R, Khanna S, Hecht G, Gerding DN. Results from a randomized, placebo-controlled clinical trial of a RBX2660-A microbiota-based drug for the prevention of recurrent Clostridium difficile infection. Clin Infect Dis 2018;67:1198-204.
44. Khanna S, Pardi DS, Kelly CR, et al. A novel microbiome therapeutic increases gut microbial diversity and prevents recurrent Clostridium difficile infection. J Infect Dis 2016;214:173-81.
45. Petrof EO, Gloor GB, Vanner SJ, et al. Stool substitute transplant therapy for the eradication of Clostridium difficile infection: 'RePOOPulating' the gut. Microbiome 2013;1:3.
46. Gerding DN, Meyer T, Lee C, et al. Administration of spores of nontoxigenic Clostridium difficile strain M3 for prevention of recurrent C. difficile infection: a randomized clinical trial. JAMA 2015;313:1719-27.