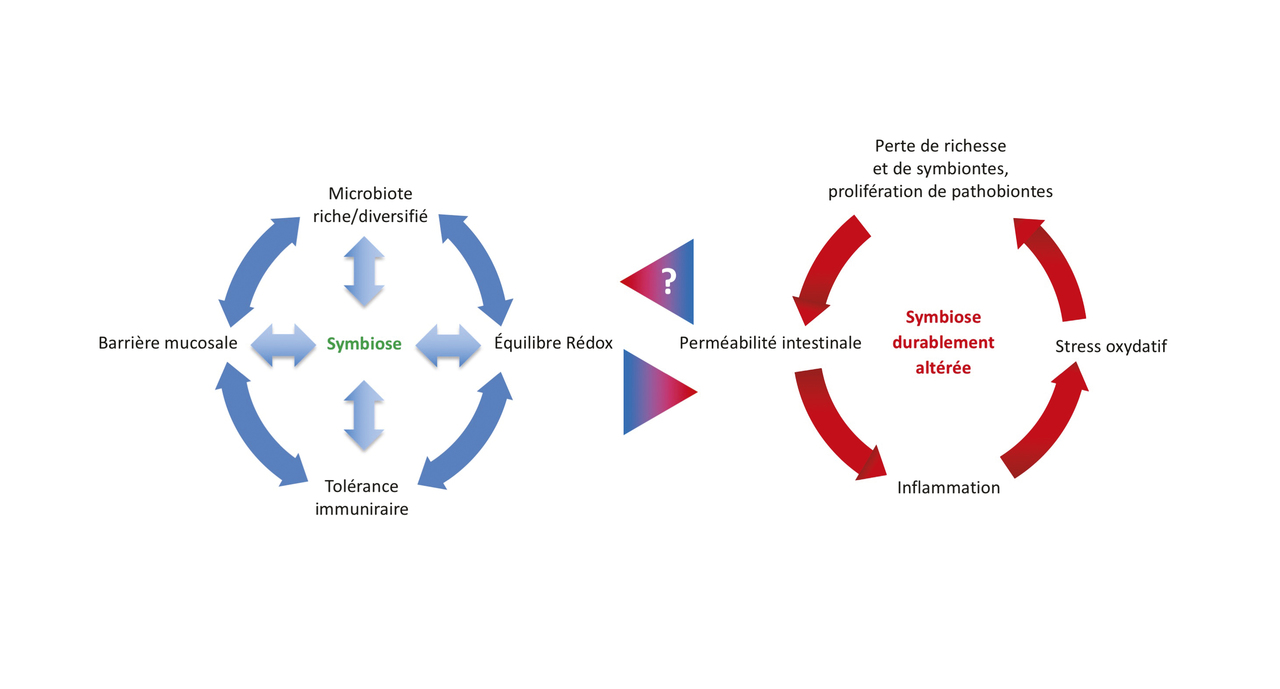
Dysbiose du microbiote et maladies immunes chroniques
Standards analytiques pour l’analyse de composition du microbiote
Altération du potentiel fonctionnel du microbiote
Dysbiose et sévérité des maladies chroniques
Une altération concomitante de paramètres du microbiote et de l’hôte (
Microbiote et obésité ou diabète
Microbiote et maladies hépatiques
Microbiote et syndrome de l’intestin irritable
Microbiote et cancers
Microbiote et maladies neuro-dégénératives ou neuropsychiatriques
Vers une médecine personnalisée ?
Métagénomique en shotgun
C’est l’approche de caractérisation moléculaire du microbiote intestinal passant par l’extraction d’ADN total et le séquençage nouvelle génération. Après « projection » sur le catalogue de tous les gènes identifiés (10 millions à ce jour), les millions de petites séquences produites permettent de générer un profil d’abondance des gènes du métagénome de chaque échantillon. Dans un second temps, le regroupement en génomes permet de générer un profil d’abondance des espèces métagénomiques. Les gènes permettent également de dresser la carte métabolique potentielle de chaque métagénome.
Qu’est-ce qu’un animal sans germes ?
Obtenus initialement par hystérectomie ou transfert d’embryons pour les mammifères ou à partir de l’œuf pour les oiseaux, les animaux sans germes ou axéniques sont élevés en enceintes stériles et en présence d’air, d’eau, de litières et d’aliments stérilisés. Ils peuvent être inoculés avec des micro-organismes choisis (souches pures, cocktails ou contenus intestinaux), ce qui permet d’en étudier le rôle fonctionnel.
2. Costea PI, Zeller G, Sunagawa S, et al. Towards standards for human fecal sample processing in metagenomics studies. Nature Biotec 2017;35:1069-77.
3. Thomas V, Clark J, Doré J. Fecal microbiota analysis: an overview of sample collection methods and sequencing strategies. Future Microbiol 2015;10:1485-504.
4. Cotillard A, Kennedy SP, Kong LC, et al. Dietary intervention impact on gut microbial richness. Nature 2013;500:585-8.
5. Le Chatelier E, Nielsen T, Qin J, et al. Richness of human gut microbiome correlates with metabolic markers. Nature 2013;500:541-6.
6. Qin N, Yang FL, Li A, et al. Alterations of the human gut microbiome in liver cirrhosis. Nature 2014;513:59-64.
7. Hartmann P, Seebauer CT, Schnabl B. Alcoholic liver disease: the gut microbiome and liver cross talk. Alcohol Clin Exp Res 2015;39:763-75.
8. Tap J, Derrien M, Tornblom H, et al. Identification of an intestinal microbiota signature associated with severity of irritable bowel syndrome. Gastroenterology 2017;152:111.
9. Van de Guchte M, Blottière HM, Doré J. Humans as holobionts: implications for prevention and therapy. Microbiome 2018;6:81.
10. Ridaura VK, Faith JJ, Rey Fe, et al. Gut microbiota from twins discordant for obesity modulate metabolism in mice. Science 2013;341:1241214.
11. Schaubeck M, Clavel T, Calasan J, et al. Dysbiotic gut microbiota causes transmissible Crohn’s disease-like ileitis independent of failure in antimicrobial defence. Gut 2016;65:225-37.
12. Le Roy T, Llopis M, Lepage P, et al. Intestinal microbiota determines development of non-alcoholic fatty liver disease in mice. Gut 2013;62:1787-94.
13. Kelly JR, Borre Y, O’ Brien C, et al. Transferring the blues: depression-associated gut microbiota induces neurobehavioural changes in the rat. J Psych Res 2016;82:109-18.
14. Zeller G, Tap J, Voigt AY, et al. Potential of fecal microbiota for early-stage detection of colorectal cancer. Mol Sys Biol 2014;10:766.
15. Routy B, Le Chatelier E, Derosa L, et al. Gut microbiome influences efficacy of PD-1–based immunotherapy against epithelial tumors. Science 2018;359:91-7.
16. Valles-Colomer M, Falony G, Darzi Y, et al. The neuroactive potential of the human gut microbiota in quality of life and depression. Nat Microbiol 2019;4:623-32.
17. Harach T, Marungruang N, Duthilleul N, et al. Reduction of Abeta amyloid pathology in APPPS1 transgenic mice in the absence of gut microbiota. SciReports 2017;7:41802.
18. Kang DW, Adams JB, Coleman DM, et al. Long-term benefits of microbiota transfer therapy on autism symptoms and gut microbiota. Sci Reports 2019;9:5821.
19. Opazo MC, Ortega-Rocha EM, Coronado-Arrázola I, et al. Intestinal microbiota influences non-intestinal related autoimmune diseases. Front Microbiol 2018;9:432.
Dans cet article
- Dysbiose du microbiote et maladies immunes chroniques
- Standards analytiques pour l’analyse de composition du microbiote
- Altération du potentiel fonctionnel du microbiote
- Dysbiose et sévérité des maladies chroniques
- Microbiote et obésité ou diabète
- Microbiote et maladies hépatiques
- Microbiote et syndrome de l’intestin irritable
- Microbiote et cancers
- Microbiote et maladies neuro-dégénératives ou neuropsychiatriques
- Vers une médecine personnalisée ?

 Encadrés
Encadrés